医療系の仕事をしています。生命の尊さ、美しさがどのようなメカニズムで生じるのかに興味があります。科学の方法論を用いて、このような問いに応えたい、私はこう思って医学生物学の基礎研究のトレーニングを受けてきました。生命を科学的手法を用いて理解を試みる上で、genomeを始めとした種々の大量データの処理が必要不可欠であることを痛感しました。また、生命科学が物理学、数学、統計学、有機化学などの種々の学問と深い関わりを持つことを実感しました。そのため、このブログは広範囲の学問領域に関しての記事を載せています。日々の学習内容を文書に書き残し、それを読み返すことによって、体系化された知識を身に付けることを目標としています。どうぞよろしくお願いします。
Rのバッチ処理で三角形の面積を計算させてみた。
$ vim triangle.R
takasa <- 30
teihen <- 4
menseki <- takasa * teihen /2
menseki
$ R CMD BATCH triangle.R triangle_R.txt
$ cat triangle_R.txt
R version 2.10.1 (2009-12-14)
Copyright (C) 2009 The R Foundation for Statistical Computing
ISBN 3-900051-07-0
Rは、自由なソフトウェアであり、「完全に無保証」です。
一定の条件に従えば、自由にこれを再配布することができます。
配布条件の詳細に関しては、'license()'あるいは'licence()'と入力してください。
Rは多くの貢献者による共同プロジェクトです。
詳しくは'contributors()'と入力してください。
また、RやRのパッケージを出版物で引用する際の形式については
'citation()'と入力してください。
'demo()'と入力すればデモをみることができます。
'help()'とすればオンラインヘルプが出ます。
'help.start()'でHTMLブラウザによるヘルプがみられます。
'q()'と入力すればRを終了します。
> takasa <- 30
> teihen <- 4
> menseki <- takasa * teihen /2
> menseki
[1] 60
>
>
> proc.time()
ユーザ システム 経過
0.460 0.052 1.309
takasa <- 30
teihen <- 4
menseki <- takasa * teihen /2
menseki
$ R CMD BATCH triangle.R triangle_R.txt
$ cat triangle_R.txt
R version 2.10.1 (2009-12-14)
Copyright (C) 2009 The R Foundation for Statistical Computing
ISBN 3-900051-07-0
Rは、自由なソフトウェアであり、「完全に無保証」です。
一定の条件に従えば、自由にこれを再配布することができます。
配布条件の詳細に関しては、'license()'あるいは'licence()'と入力してください。
Rは多くの貢献者による共同プロジェクトです。
詳しくは'contributors()'と入力してください。
また、RやRのパッケージを出版物で引用する際の形式については
'citation()'と入力してください。
'demo()'と入力すればデモをみることができます。
'help()'とすればオンラインヘルプが出ます。
'help.start()'でHTMLブラウザによるヘルプがみられます。
'q()'と入力すればRを終了します。
> takasa <- 30
> teihen <- 4
> menseki <- takasa * teihen /2
> menseki
[1] 60
>
>
> proc.time()
ユーザ システム 経過
0.460 0.052 1.309
Cで変数の格納
$ vim practice2.c
a
#include <stdio.h>
int main(void)
{
int suti1, suti2, suti3;
suti1 = 10;
suti2 = 20;
suti3 = 30;
printf("suti1の値は%dです。suti2値は%dです。suti3の値は%dです\n", suti1, suti2, suti3);
return 0;
}
Esc
:wq
$ gcc -o practice2 practice2.c
$ ./practice2
suti1の値は10です。suti2値は20です。suti3の値は30です
int型変数suti1,suti2,suti3を宣言してから、各々に整数を格納し、print関数によって表示させた。
a
#include <stdio.h>
int main(void)
{
int suti1, suti2, suti3;
suti1 = 10;
suti2 = 20;
suti3 = 30;
printf("suti1の値は%dです。suti2値は%dです。suti3の値は%dです\n", suti1, suti2, suti3);
return 0;
}
Esc
:wq
$ gcc -o practice2 practice2.c
$ ./practice2
suti1の値は10です。suti2値は20です。suti3の値は30です
int型変数suti1,suti2,suti3を宣言してから、各々に整数を格納し、print関数によって表示させた。
私の専攻はbioinformaticsです。(C language)
$ vim practice1.c
a
#include<stdio.h>
int main(void)
{
printf("私の専攻は\n");
printf("Bioinformaticsです\n");
return 0;
}
Esc
:wq
$ gcc -o practice1 practice1.c
$ ./practice1
私の専攻は
Bioinformaticsです
a
#include<stdio.h>
int main(void)
{
printf("私の専攻は\n");
printf("Bioinformaticsです\n");
return 0;
}
Esc
:wq
$ gcc -o practice1 practice1.c
$ ./practice1
私の専攻は
Bioinformaticsです
正規分布に従う乱数の作成及びプロット。シミュレーション。
$ R
install.packages("scatterplot3d")
library(scatterplot3d)
a <- rnorm(10) # 正規分布に従う乱数を10個生成
b <- rnorm(10) # 正規分布に従う乱数を10個生成
c <- rnorm(10) # 正規分布に従う乱数を10個生成
d <- rnorm(100) # 正規分布に従う乱数を100個生成
e <- rnorm(100) # 正規分布に従う乱数を100個生成
f <- rnorm(100) # 正規分布に従う乱数を100個生成
g <- rnorm(1000) # 正規分布に従う乱数を1000個生成
h <- rnorm(1000) # 正規分布に従う乱数を1000個生成
i <- rnorm(1000) # 正規分布に従う乱数を1000個生成
x <- rnorm(10000) # 正規分布に従う乱数を10000個生成
y <- rnorm(10000) # 正規分布に従う乱数を10000個生成
z <- rnorm(10000) # 正規分布に従う乱数を10000個生成
png("110217.rnorm.png")
par(mfrow = c(2,2))
scatterplot3d(a,b,c)
scatterplot3d(d,e,f)
scatterplot3d(g,h,i)
scatterplot3d(x,y,z)
dev.off()
う~ん。胸が熱くなる。
install.packages("scatterplot3d")
library(scatterplot3d)
a <- rnorm(10) # 正規分布に従う乱数を10個生成
b <- rnorm(10) # 正規分布に従う乱数を10個生成
c <- rnorm(10) # 正規分布に従う乱数を10個生成
d <- rnorm(100) # 正規分布に従う乱数を100個生成
e <- rnorm(100) # 正規分布に従う乱数を100個生成
f <- rnorm(100) # 正規分布に従う乱数を100個生成
g <- rnorm(1000) # 正規分布に従う乱数を1000個生成
h <- rnorm(1000) # 正規分布に従う乱数を1000個生成
i <- rnorm(1000) # 正規分布に従う乱数を1000個生成
x <- rnorm(10000) # 正規分布に従う乱数を10000個生成
y <- rnorm(10000) # 正規分布に従う乱数を10000個生成
z <- rnorm(10000) # 正規分布に従う乱数を10000個生成
png("110217.rnorm.png")
par(mfrow = c(2,2))
scatterplot3d(a,b,c)
scatterplot3d(d,e,f)
scatterplot3d(g,h,i)
scatterplot3d(x,y,z)
dev.off()
う~ん。胸が熱くなる。
scatterplod3dで3次元散布図を作成した★
$ R
install.packages("scatterplot3d")
library(scatterplot3d)
x <- c(1,2,3,4,5,6,7,8,9)
y <- c(9,8,7,6,5,4,3,2,1)
z <- c(21,22,23,24,25,26,27,28,29)
png("110217.scatterplot3d.png")
scatterplot3d(x,y,z)
dev.off()
install.packages("scatterplot3d")
library(scatterplot3d)
x <- c(1,2,3,4,5,6,7,8,9)
y <- c(9,8,7,6,5,4,3,2,1)
z <- c(21,22,23,24,25,26,27,28,29)
png("110217.scatterplot3d.png")
scatterplot3d(x,y,z)
dev.off()
perlで塩基配列を扱ってみたよん(^^)
perlを用いて、DNAの塩基配列を出力したり、二つのDNAフラグメントを結合したりしてみました(^_^)
$ vim 110215.1.pl
a
use strict;
use warnings;
#Storing DNA in a variable, and printing it
#First we store the DNA in a variable called $DNA
my $DNA = 'AGCGGAGGACGGGAAAATTACTACGGCATTAGC';
# Next, we print the DNA onto the screen
print "$DNA\n";
Esc
:wq
$ perl 110215.1.pl
AGCGGAGGACGGGAAAATTACTACGGCATTAGC
$ vim 110215.2.pl
a
1 use strict;
2
3 #Store two DNA fragments into two vairiables called $DAN1 and $DNA2
4 my $DNA1 = 'ACGGGAGGACGGGAAAATTACTACGGCATTAGC';
5 my $DNA2 = 'ATAGTGCCGTGAGAGTGATGTAGTA';
6
7 # Print the DNA onto the screen
8 print "Here are the original two DNA fragments:\n\n";
9 print "DNA1 is $DNA1\n";
10 print "DNA2 is $DNA2\n\n";
11
12 #Concatenate the DNA fragments into a third variable and print them
13 #Using #string interpolation"
14 my $DNA3 = "$DNA1$DNA2";
15
16 print "Here is the concatenation of the first two fragments(version1):\n\n";
17 print "$DNA3\n\n";
18
19 #An alternative way using the "dot operator";
20 #Concatenate the DNA fragments into a third variable and print them
21 my $DNA3 = $DNA1 . $DNA2;
22
23 print "Here is the concatenation of the first two fragments(version2):\n";
24 print "$DNA3\n\n";
25
26 #print the same thing without using the variable $DNA3
27 print "Here is the concatenation of the first two fragments(version3):\n";
28 print $DNA1, $DNA2, "\n";
29
30
Esc
$ perl 110215.2.pl
Here are the original two DNA fragments:
DNA1 is ACGGGAGGACGGGAAAATTACTACGGCATTAGC
DNA2 is ATAGTGCCGTGAGAGTGATGTAGTA
Here is the concatenation of the first two fragments(version1):
ACGGGAGGACGGGAAAATTACTACGGCATTAGCATAGTGCCGTGAGAGTGATGTAGTA
Here is the concatenation of the first two fragments(version2):
ACGGGAGGACGGGAAAATTACTACGGCATTAGCATAGTGCCGTGAGAGTGATGTAGTA
Here is the concatenation of the first two fragments(version3):
ACGGGAGGACGGGAAAATTACTACGGCATTAGCATAGTGCCGTGAGAGTGATGTAGTA
$ vim 110215.1.pl
a
use strict;
use warnings;
#Storing DNA in a variable, and printing it
#First we store the DNA in a variable called $DNA
my $DNA = 'AGCGGAGGACGGGAAAATTACTACGGCATTAGC';
# Next, we print the DNA onto the screen
print "$DNA\n";
Esc
:wq
$ perl 110215.1.pl
AGCGGAGGACGGGAAAATTACTACGGCATTAGC
$ vim 110215.2.pl
a
1 use strict;
2
3 #Store two DNA fragments into two vairiables called $DAN1 and $DNA2
4 my $DNA1 = 'ACGGGAGGACGGGAAAATTACTACGGCATTAGC';
5 my $DNA2 = 'ATAGTGCCGTGAGAGTGATGTAGTA';
6
7 # Print the DNA onto the screen
8 print "Here are the original two DNA fragments:\n\n";
9 print "DNA1 is $DNA1\n";
10 print "DNA2 is $DNA2\n\n";
11
12 #Concatenate the DNA fragments into a third variable and print them
13 #Using #string interpolation"
14 my $DNA3 = "$DNA1$DNA2";
15
16 print "Here is the concatenation of the first two fragments(version1):\n\n";
17 print "$DNA3\n\n";
18
19 #An alternative way using the "dot operator";
20 #Concatenate the DNA fragments into a third variable and print them
21 my $DNA3 = $DNA1 . $DNA2;
22
23 print "Here is the concatenation of the first two fragments(version2):\n";
24 print "$DNA3\n\n";
25
26 #print the same thing without using the variable $DNA3
27 print "Here is the concatenation of the first two fragments(version3):\n";
28 print $DNA1, $DNA2, "\n";
29
30
Esc
$ perl 110215.2.pl
Here are the original two DNA fragments:
DNA1 is ACGGGAGGACGGGAAAATTACTACGGCATTAGC
DNA2 is ATAGTGCCGTGAGAGTGATGTAGTA
Here is the concatenation of the first two fragments(version1):
ACGGGAGGACGGGAAAATTACTACGGCATTAGCATAGTGCCGTGAGAGTGATGTAGTA
Here is the concatenation of the first two fragments(version2):
ACGGGAGGACGGGAAAATTACTACGGCATTAGCATAGTGCCGTGAGAGTGATGTAGTA
Here is the concatenation of the first two fragments(version3):
ACGGGAGGACGGGAAAATTACTACGGCATTAGCATAGTGCCGTGAGAGTGATGTAGTA
BMI, MCV, MCH, MCHCを計算&正常判定をRでやってみた。
患者さんの検査値が以下のようだとします。
Wight 160cm
Height 47kg
WBC 6800/ul
RBC 412*10^4/ul
Hct 38.8%
Hb 11.8g/dl
$ R
Height <- 160*10^-2
Weight <- 47
BMI <- Weight / Height^2 #Body Mass index
BMI
[1] 18.35937 #22ぐらいが丁度いいと言われている。
WBC <- 6800 #white blood cell.白血球。
RBC <- 4.12 * 10^6 # Red blood cell。赤血球。
Hct <- 38.8 #Hct:ヘマトクリット。全血二体する赤血球容積。
Hb <- 11.8 #ヘモグロビンの濃度。
(4300<WBC) && (WBC<6900)
[1] TRUE
(446*10^4<RBC) && (RBC<515*10^4)
[1] FALSE
(43.2<Hct) && (Hct<48.6)
[1] FALSE
MCV <- Hct*10*10^6/RBC #平均赤血球容積
MCV
[1] 94.17476
(91.3<MCV) && (MCV<99.3)
[1] TRUE
MCH <- Hb*10*10^6/RBC #平均赤血球ヘモグロビン量
MCH
[1] 28.64078
(30.0<MCH) && (MCH<32.7)
[1] FALSE
MCHC <- Hb*100/Hct #平均赤血球ヘモグロビン濃度
MCHC
[1] 30.41237
(32.3<MCHC) && (MCHC<33.7)
[1] FALSE
Wight 160cm
Height 47kg
WBC 6800/ul
RBC 412*10^4/ul
Hct 38.8%
Hb 11.8g/dl
$ R
Height <- 160*10^-2
Weight <- 47
BMI <- Weight / Height^2 #Body Mass index
BMI
[1] 18.35937 #22ぐらいが丁度いいと言われている。
WBC <- 6800 #white blood cell.白血球。
RBC <- 4.12 * 10^6 # Red blood cell。赤血球。
Hct <- 38.8 #Hct:ヘマトクリット。全血二体する赤血球容積。
Hb <- 11.8 #ヘモグロビンの濃度。
(4300<WBC) && (WBC<6900)
[1] TRUE
(446*10^4<RBC) && (RBC<515*10^4)
[1] FALSE
(43.2<Hct) && (Hct<48.6)
[1] FALSE
MCV <- Hct*10*10^6/RBC #平均赤血球容積
MCV
[1] 94.17476
(91.3<MCV) && (MCV<99.3)
[1] TRUE
MCH <- Hb*10*10^6/RBC #平均赤血球ヘモグロビン量
MCH
[1] 28.64078
(30.0<MCH) && (MCH<32.7)
[1] FALSE
MCHC <- Hb*100/Hct #平均赤血球ヘモグロビン濃度
MCHC
[1] 30.41237
(32.3<MCHC) && (MCHC<33.7)
[1] FALSE
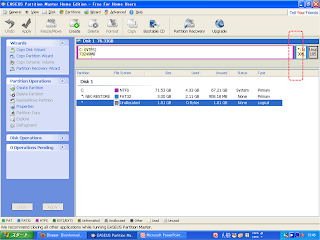
Windows XPで未割り当て領域にCドライブを拡張してみた!!
” EASEUS Partition Manager Home Edit ”
は結構すごいですよ!!
なんといっても、マウスを使ってグニャッと動かすだけでパーティションの両々を変更できちゃう!!
http://www.partition-tool.com/personal.htm
にいって
EASEUS Partition Manager Home Editを立ち上げてみます。
赤い波線で囲ったパーティションの間のエッジをグイグイ動かせばパーティションの容量を自由自在に変更することができます♪
最後は、applyをクリックすれば変更が反映されました★☆
は結構すごいですよ!!
なんといっても、マウスを使ってグニャッと動かすだけでパーティションの両々を変更できちゃう!!
http://www.partition-tool.com/personal.htm
にいって
解凍は、Lhasaみたいな解凍ソフトで一瞬で解凍できます。
中にインストーラー入ってるんで、さくさくインストールします。
インストールしたら、実際に
EASEUS Partition Manager Home Editを立ち上げてみます。
赤い波線で囲ったパーティションの間のエッジをグイグイ動かせばパーティションの容量を自由自在に変更することができます♪
最後は、applyをクリックすれば変更が反映されました★☆
Windows環境のRで環境変数をいじってRgraphvizのインストールに成功したよ!!♪
http://www.graphviz.org/pub/graphviz/stable/windows/
ここで
graphviz-2.20.3a.msi
をダウンロードしてインストール!!!
次に、
コントロールパネル -> システム -> 詳細設定タブ -> 環境変数(N)
すると以下のウィンドウが開く。
ここで、下段のシステム環境変数(S):赤線でかこってあるやつ:をクリック。
変数名 Path
変数値 C:\Program Files\Graphviz2.20\bin\
を入力。
あとは、2回分OKを押す♪
一度、パソコンを再起動かける♪再起動しないとうまくいかなかったわ(涙)
これで、Windows XPに対する設定は終わり!
最後にRを開いて、パッケージ Rgraphvizをインストールしてみる☆
source("http://bioconductor.org/biocLite.R")
biocLite("Rgraphviz")
library("Rgraphviz")
要求されたパッケージ Biobase をロード中です
Welcome to Bioconductor
Vignettes contain introductory material. To view, type
'openVignette()'. To cite Bioconductor, see
'citation("Biobase")' and for packages 'citation(pkgname)'.
要求されたパッケージ Category をロード中です
要求されたパッケージ AnnotationDbi をロード中です
要求されたパッケージ DBI をロード中です
# キタ━━━(゚(゚∀(゚∀゚(☆∀☆)゚∀゚)∀゚)゚)━━━!!!
#動作テストしてみます。
set.seed(123)
V <- letters[1:10]
M <- 1:4
graph <- randomGraph(V, M, 0.2)
graph <- layoutGraph(graph)
png("110214.graph.png")
renderGraph(graph)
dev.off()
ここで
graphviz-2.20.3a.msi
をダウンロードしてインストール!!!
次に、
コントロールパネル -> システム -> 詳細設定タブ -> 環境変数(N)
すると以下のウィンドウが開く。
ここで、下段のシステム環境変数(S):赤線でかこってあるやつ:をクリック。
変数名 Path
変数値 C:\Program Files\Graphviz2.20\bin\
を入力。
あとは、2回分OKを押す♪
一度、パソコンを再起動かける♪再起動しないとうまくいかなかったわ(涙)
これで、Windows XPに対する設定は終わり!
最後にRを開いて、パッケージ Rgraphvizをインストールしてみる☆
source("http://bioconductor.org/biocLite.R")
biocLite("Rgraphviz")
library("Rgraphviz")
要求されたパッケージ Biobase をロード中です
Welcome to Bioconductor
Vignettes contain introductory material. To view, type
'openVignette()'. To cite Bioconductor, see
'citation("Biobase")' and for packages 'citation(pkgname)'.
要求されたパッケージ Category をロード中です
要求されたパッケージ AnnotationDbi をロード中です
要求されたパッケージ DBI をロード中です
# キタ━━━(゚(゚∀(゚∀゚(☆∀☆)゚∀゚)∀゚)゚)━━━!!!
#動作テストしてみます。
set.seed(123)
V <- letters[1:10]
M <- 1:4
graph <- randomGraph(V, M, 0.2)
graph <- layoutGraph(graph)
png("110214.graph.png")
renderGraph(graph)
dev.off()
Gene ontologyのデータをGO.dbで取得したのちネットワークを描かせた♪♪
GO, itself, is a structured terminology. The ontology describes genes and gene products and is divided into three separate ontologies. One for cellular component (CC),one for molecular function (MF) and one for biological process (BP).
つまり、
BPは、生物学的プロセス
MFは、分子機能
CCは、細胞の構成単位
っていうことみたいね。
これらのGOのデータベースを覗いて見ようと思う。
install.packages("igraph")
source("http://bioconductor.org/biocLite.R")
biocLite("GO.db")
library(GO.db)
library(igraph)
BP <- toTable(GOBPPARENTS)
CC <- toTable(GOCCPARENTS)
MF <- toTable(GOMFPARENTS)
#これで、BP(biological process)と、CC(Celular Component)と、MF(Molecular Function)をデータテーブルにできた!!!
#そしたら、これらを覗いてみよう♪
BP[1,]
go_id go_id RelationshipType
1 GO:0000001 GO:0048308 isa
CC[1,]
go_id go_id RelationshipType
1 GO:0000015 GO:0043234 isa
MF[1,]
go_id go_id RelationshipType
1 GO:0051082 GO:0005515 isa
#たとえば、BPの1から1000行までの一列目と二列目を抜き出してオブジェクトBP2を生成すると
BP2 <- BP[1:1000, 1:2]
BP2[1,]
go_id go_id.1
1 GO:0000001 GO:0048308
class(BP2)
[1] "data.frame"
BP3 <- as.matrix(BP2)
#ここで、BP3オブジェクトは、辺リストとみなすことができるので、graph.edgelist(辺リストの行列)により、igraphで利用可能なgraphオブジェクトを生成することができる♪
g.BP3 <- graph.edgelist(BP3)
plot(g.BP3, vertex.size = 1, vertex.color = 5, edge.color = 7, edge.arrow.size = 0.1 vertex.label.cex = 0.001, )
#vertex.size :頂点の大きさ。デフォルトは15。vertex.color:頂点の色。vertex.label.cex:頂点のラベルの大きさ.edge.arrow.size:矢印の大きさ。デフォルトは1。
png("test.png")
plot(g.BP3, vertex.size = 3, vertex.color = "yellow", edge.color = "green", edge.arrow.size = 0.01, vertex.label.cex = 0.1)
dev.off()
つまり、
BPは、生物学的プロセス
MFは、分子機能
CCは、細胞の構成単位
っていうことみたいね。
これらのGOのデータベースを覗いて見ようと思う。
install.packages("igraph")
source("http://bioconductor.org/biocLite.R")
biocLite("GO.db")
library(GO.db)
library(igraph)
BP <- toTable(GOBPPARENTS)
CC <- toTable(GOCCPARENTS)
MF <- toTable(GOMFPARENTS)
#これで、BP(biological process)と、CC(Celular Component)と、MF(Molecular Function)をデータテーブルにできた!!!
#そしたら、これらを覗いてみよう♪
BP[1,]
go_id go_id RelationshipType
1 GO:0000001 GO:0048308 isa
CC[1,]
go_id go_id RelationshipType
1 GO:0000015 GO:0043234 isa
MF[1,]
go_id go_id RelationshipType
1 GO:0051082 GO:0005515 isa
#たとえば、BPの1から1000行までの一列目と二列目を抜き出してオブジェクトBP2を生成すると
BP2 <- BP[1:1000, 1:2]
BP2[1,]
go_id go_id.1
1 GO:0000001 GO:0048308
class(BP2)
[1] "data.frame"
BP3 <- as.matrix(BP2)
#ここで、BP3オブジェクトは、辺リストとみなすことができるので、graph.edgelist(辺リストの行列)により、igraphで利用可能なgraphオブジェクトを生成することができる♪
g.BP3 <- graph.edgelist(BP3)
plot(g.BP3, vertex.size = 1, vertex.color = 5, edge.color = 7, edge.arrow.size = 0.1 vertex.label.cex = 0.001, )
#vertex.size :頂点の大きさ。デフォルトは15。vertex.color:頂点の色。vertex.label.cex:頂点のラベルの大きさ.edge.arrow.size:矢印の大きさ。デフォルトは1。
png("test.png")
plot(g.BP3, vertex.size = 3, vertex.color = "yellow", edge.color = "green", edge.arrow.size = 0.01, vertex.label.cex = 0.1)
dev.off()
igraphでランダムグラフの作成♪♪
ランダムグラフとは、グラフに含まれる2つの頂点の間に一定の確率で辺を張ることによって作られるグラフである。ランダムグラフについて最初に体系的な研究をまとめた2人の数学者の名前から、Erdos-Renyi graphとも呼ばれる。
random.graph.game()の引数は
頂点数
2頂点間で辺を張る確率p
無向グラフ(デフォルト)か有向グラフかを決める。
例えば、
$ R
library(igraph)
g1 <- random.graph.game(50, p=0.01)
g2 <- random.graph.game(50, p=0.01, directed = TRUE)
g3 <- random.graph.game(50, p=0.1)
g4 <- random.graph.game(50, p=0.1, directed = TRUE)
png("110214.random.graph.game.png")
par(mfrow = c(2,2))
plot(g1)
plot(g2)
plot(g3)
plot(g4)
dev.off()
random.graph.game()の引数は
頂点数
2頂点間で辺を張る確率p
無向グラフ(デフォルト)か有向グラフかを決める。
例えば、
$ R
library(igraph)
g1 <- random.graph.game(50, p=0.01)
g2 <- random.graph.game(50, p=0.01, directed = TRUE)
g3 <- random.graph.game(50, p=0.1)
g4 <- random.graph.game(50, p=0.1, directed = TRUE)
png("110214.random.graph.game.png")
par(mfrow = c(2,2))
plot(g1)
plot(g2)
plot(g3)
plot(g4)
dev.off()
igraphで無向グラフを描写する。
#無向グラフを作成する
e.matrix <- matrix(c(1,2,1,3,1,4,2,3),ncol =2 , byrow = TRUE)
e.matrix
[,1] [,2]
[1,] 1 2
[2,] 1 3
[3,] 1 4
[4,] 2 3
g4 <- graph.edgelist(e.matrix-1, directed = FALSE)
g4
Vertices: 4
Edges: 4
Directed: FALSE
Edges:
[0] 0 -- 1
[1] 0 -- 2
[2] 0 -- 3
[3] 1 -- 2
png("110214.g4.png")
plot(g4)
dev.off()
e.matrix <- matrix(c(1,2,1,3,1,4,2,3),ncol =2 , byrow = TRUE)
e.matrix
[,1] [,2]
[1,] 1 2
[2,] 1 3
[3,] 1 4
[4,] 2 3
g4 <- graph.edgelist(e.matrix-1, directed = FALSE)
g4
Vertices: 4
Edges: 4
Directed: FALSE
Edges:
[0] 0 -- 1
[1] 0 -- 2
[2] 0 -- 3
[3] 1 -- 2
png("110214.g4.png")
plot(g4)
dev.off()
辺リストを与えて有向グラフを描く♪
#ベクトルとして辺リストを作成する。
e.list <- c(1,2,1,3,2,1,2,3,4,1)
#行列として辺リストを作成する。
e.matrix <- matrix(e.list, ncol = 2, byrow = TRUE)
#このbyrow=TRUEのテクニックは必見・・・。普通かな笑
e.list
[1] 1 2 1 3 2 1 2 3 4 1
e.matrix
[,1] [,2]
[1,] 1 2
[2,] 1 3
[3,] 2 1
[4,] 2 3
[5,] 4 1
e.list-1
[1] 0 1 0 2 1 0 1 2 3 0
g2 <- graph(e.list-1, n=4)
e.matrix-1
[,1] [,2]
[1,] 0 1
[2,] 0 2
[3,] 1 0
[4,] 1 2
[5,] 3 0
g3 <- graph.edgelist(e.matrix - 1)
png("110214.g23.png")
par(mfrow = c(1,2))
plot(g2)
plot(g3)
dev.off()
e.list <- c(1,2,1,3,2,1,2,3,4,1)
#行列として辺リストを作成する。
e.matrix <- matrix(e.list, ncol = 2, byrow = TRUE)
#このbyrow=TRUEのテクニックは必見・・・。普通かな笑
e.list
[1] 1 2 1 3 2 1 2 3 4 1
e.matrix
[,1] [,2]
[1,] 1 2
[2,] 1 3
[3,] 2 1
[4,] 2 3
[5,] 4 1
e.list-1
[1] 0 1 0 2 1 0 1 2 3 0
g2 <- graph(e.list-1, n=4)
e.matrix-1
[,1] [,2]
[1,] 0 1
[2,] 0 2
[3,] 1 0
[4,] 1 2
[5,] 3 0
g3 <- graph.edgelist(e.matrix - 1)
png("110214.g23.png")
par(mfrow = c(1,2))
plot(g2)
plot(g3)
dev.off()
igraphで隣接行列を与えて有向グラフを作成してみた!!
$ R
library(igraph)
dg1 <- matrix(c(0,1,1,0,1,0,1,0,0,0,0,0,1,0,0,0), nrow=4, ncol = 4, byrow = TRUE)
dg1
[,1] [,2] [,3] [,4]
[1,] 0 1 1 0
[2,] 1 0 1 0
[3,] 0 0 0 0
[4,] 1 0 0 0
class(dg1)
[1] "matrix"
colnames(dg1) <- 1:4
rownames(dg1) <- 1:4
dg1
1 2 3 4
1 0 1 1 0
2 1 0 1 0
3 0 0 0 0
4 1 0 0 0
#igraphのパッケージでは、隣接行列をそのままネットワークデーとして用いることができないので、隣接行列からigraphで利用可能なigraphオブジェクトを生成する必要がある。
g1 <- graph.adjacency(dg1)
#adjacencyとは”隣接”という意味!
g1
Vertices: 4
Edges: 5
Directed: TRUE
Edges:
[0] '1' -> '2'
[1] '1' -> '3'
[2] '2' -> '1'
[3] '2' -> '3'
[4] '4' -> '1'
class(g1)
[1] "igraph"
png("110214.g1.png")
plot.igraph(g1)
dev.off()
library(igraph)
dg1 <- matrix(c(0,1,1,0,1,0,1,0,0,0,0,0,1,0,0,0), nrow=4, ncol = 4, byrow = TRUE)
dg1
[,1] [,2] [,3] [,4]
[1,] 0 1 1 0
[2,] 1 0 1 0
[3,] 0 0 0 0
[4,] 1 0 0 0
class(dg1)
[1] "matrix"
colnames(dg1) <- 1:4
rownames(dg1) <- 1:4
dg1
1 2 3 4
1 0 1 1 0
2 1 0 1 0
3 0 0 0 0
4 1 0 0 0
#igraphのパッケージでは、隣接行列をそのままネットワークデーとして用いることができないので、隣接行列からigraphで利用可能なigraphオブジェクトを生成する必要がある。
g1 <- graph.adjacency(dg1)
#adjacencyとは”隣接”という意味!
g1
Vertices: 4
Edges: 5
Directed: TRUE
Edges:
[0] '1' -> '2'
[1] '1' -> '3'
[2] '2' -> '1'
[3] '2' -> '3'
[4] '4' -> '1'
class(g1)
[1] "igraph"
png("110214.g1.png")
plot.igraph(g1)
dev.off()
bioperlを使ってて、塩基配列のアミノ酸への翻訳に成功した!キタ――o(・∀・`o)(o`・∀・´o)(o´・∀・)o キタ――♪
ちょっと時期早尚な感は否めないが、bioperlを用いてプログラミングを書いて、入力した塩基配列をアミノ酸に翻訳させてみた!!!!
$ sudo apt-get install bioperl
$ vim 110213.5.pl
a
use strict;
use warnings;
use Bio::Seq;
$naseq = Bio::Seq->new(-seq=>$ARGV[0]);
$aaseq = $naseq->translate();
print $aaseq->seq(), "\n";
Esc
$ perl 110213.5.pl "atgcatgcatgcatgc"
MHACM
キタ――o(・∀・`o)(o`・∀・´o)(o´・∀・)o キタ――♪
キタキタ━(ノ゚∀゚)ノ ┫:。・:*:・゚'★,。・:*:♪・゚'☆━━━!!!
$ sudo apt-get install bioperl
$ vim 110213.5.pl
a
use strict;
use warnings;
use Bio::Seq;
$naseq = Bio::Seq->new(-seq=>$ARGV[0]);
$aaseq = $naseq->translate();
print $aaseq->seq(), "\n";
Esc
$ perl 110213.5.pl "atgcatgcatgcatgc"
MHACM
キタ――o(・∀・`o)(o`・∀・´o)(o´・∀・)o キタ――♪
キタキタ━(ノ゚∀゚)ノ ┫:。・:*:・゚'★,。・:*:♪・゚'☆━━━!!!
Perlで条件文
$ vim 110213.pl
a
use strict;
use warnings;
print "好きな遺伝子を選んでください。\n";
print "1 = capase\n";
print "2 = hox\n";
print "3 = rhodopsin\n";
my $line = <STDIN>; # ユーザから1行入力
if ($line == 1) { # 1番か?
print "アポトーシスの実行に関与します。\n";
} elsif ($line == 2) { # 2番か?
print "発生時の体節の形勢に関与します。\n";
} elsif ($line == 3) { # 3番か?
print "視細胞の働きを助けます\n";
} else { # それ以外か?
print "ちゃんと言われた遺伝子を選んでくださいまし。\n";
}
Esc
:wq
$ perl 110213.pl
好きな遺伝子を選んでください。
1 = capase
2 = hox
3 = rhodopsin
1
アポトーシスの実行に関与します。
$ perl 110213.pl
好きな遺伝子を選んでください。
1 = capase
2 = hox
3 = rhodopsin
2
発生時の体節の形勢に関与します。
$ perl 110213.pl
好きな遺伝子を選んでください。
1 = capase
2 = hox
3 = rhodopsin
3
視細胞の働きを助けます
$ perl 110213.pl
好きな遺伝子を選んでください。
1 = capase
2 = hox
3 = rhodopsin
4
ちゃんと言われた遺伝子を選んでくださいまし。
$ vim 110213.2.pl
a
use strict;
use warnings;
print "ヒトの染色体の本数を述べなさい。\n";
my $line = <STDIN>; # ユーザから1行入力
if ($line == "46") {
print "その通り!!\n";
} else {
print "違います。\n";
}
Esc
:wq
$ perl 110213.2.pl
ヒトの染色体の本数を述べなさい。
40
違います。
$ perl 110213.2.pl
ヒトの染色体の本数を述べなさい。
46
その通り!!
kappa@kappa-desktop:~/2011/perl$
$ vim 110213.3.pl
a
print "あなたの身長を入力してください。\n";
my $x = 171;
my $line = <STDIN>;
if ($x > $line) {
print "あなたの身長$lineは日本人男性の平均身長よりも小さいです\n";
} else {
print "あなたの身長$lineは日本人男性の平均身長よりも大きいです\n";
}
Esc
:wq
$ perl 110213.3.pl
あなたの身長を入力してください。
168
あなたの身長168
は日本人男性の平均身長よりも小さいです
$ perl 110213.3.pl
あなたの身長を入力してください。
172
あなたの身長172
は日本人男性の平均身長よりも大きいです
$ vim 110214.4.pl
a
use strict;
use warnings;
print "ただいまの時刻を入力してちょ。\n";
my $line = <STDIN>;
if ($line < 8) {
print "おはよ!\n";
} elsif ($line == 12) {
print "おなかすいたっさ。\n";
} elsif ($line < 17) {
print "こんにちは!\n";
} else {
print "こんばんは。";
}
Esc
$ perl 110213.4.pl
ただいまの時刻を入力してちょ。
16
こんにちは!
a
use strict;
use warnings;
print "好きな遺伝子を選んでください。\n";
print "1 = capase\n";
print "2 = hox\n";
print "3 = rhodopsin\n";
my $line = <STDIN>; # ユーザから1行入力
if ($line == 1) { # 1番か?
print "アポトーシスの実行に関与します。\n";
} elsif ($line == 2) { # 2番か?
print "発生時の体節の形勢に関与します。\n";
} elsif ($line == 3) { # 3番か?
print "視細胞の働きを助けます\n";
} else { # それ以外か?
print "ちゃんと言われた遺伝子を選んでくださいまし。\n";
}
Esc
:wq
$ perl 110213.pl
好きな遺伝子を選んでください。
1 = capase
2 = hox
3 = rhodopsin
1
アポトーシスの実行に関与します。
$ perl 110213.pl
好きな遺伝子を選んでください。
1 = capase
2 = hox
3 = rhodopsin
2
発生時の体節の形勢に関与します。
$ perl 110213.pl
好きな遺伝子を選んでください。
1 = capase
2 = hox
3 = rhodopsin
3
視細胞の働きを助けます
$ perl 110213.pl
好きな遺伝子を選んでください。
1 = capase
2 = hox
3 = rhodopsin
4
ちゃんと言われた遺伝子を選んでくださいまし。
$ vim 110213.2.pl
a
use strict;
use warnings;
print "ヒトの染色体の本数を述べなさい。\n";
my $line = <STDIN>; # ユーザから1行入力
if ($line == "46") {
print "その通り!!\n";
} else {
print "違います。\n";
}
Esc
:wq
$ perl 110213.2.pl
ヒトの染色体の本数を述べなさい。
40
違います。
$ perl 110213.2.pl
ヒトの染色体の本数を述べなさい。
46
その通り!!
kappa@kappa-desktop:~/2011/perl$
$ vim 110213.3.pl
a
print "あなたの身長を入力してください。\n";
my $x = 171;
my $line = <STDIN>;
if ($x > $line) {
print "あなたの身長$lineは日本人男性の平均身長よりも小さいです\n";
} else {
print "あなたの身長$lineは日本人男性の平均身長よりも大きいです\n";
}
Esc
:wq
$ perl 110213.3.pl
あなたの身長を入力してください。
168
あなたの身長168
は日本人男性の平均身長よりも小さいです
$ perl 110213.3.pl
あなたの身長を入力してください。
172
あなたの身長172
は日本人男性の平均身長よりも大きいです
$ vim 110214.4.pl
a
use strict;
use warnings;
print "ただいまの時刻を入力してちょ。\n";
my $line = <STDIN>;
if ($line < 8) {
print "おはよ!\n";
} elsif ($line == 12) {
print "おなかすいたっさ。\n";
} elsif ($line < 17) {
print "こんにちは!\n";
} else {
print "こんばんは。";
}
Esc
$ perl 110213.4.pl
ただいまの時刻を入力してちょ。
16
こんにちは!
Rで数式を描写してみた♪高校時代を思い出す絵になった☆
png("110212_graphics.png")
x <- seq(-5,5,length=10001)
plot(c(-5,5),c(-100,100),type="n", main = "ごく簡単な関数")
par(pch=".")
points(x,x, col = "black")
points(x,x^2, col="red")
points(x,x^3, col ="yellow")
points(x,-x,col = "green")
points(x, x^2-x , col = "blue")
points(x, x^5-x^4+x^3 , col = "grey")
dev.off()
x <- seq(-5,5,length=10001)
plot(c(-5,5),c(-100,100),type="n", main = "ごく簡単な関数")
par(pch=".")
points(x,x, col = "black")
points(x,x^2, col="red")
points(x,x^3, col ="yellow")
points(x,-x,col = "green")
points(x, x^2-x , col = "blue")
points(x, x^5-x^4+x^3 , col = "grey")
dev.off()
igraph(R)によりランダムネットワークを書いてみた!!!!
$ R
install.packages("igraph")
library(igraph)
# 乱数から成る100×100のデータ行列を作成
mat <- matrix(runif(10000), nr=100)
png("110212_graph.png")
par(mfrow=c(2,1))
plot(graph.adjacency(mat>=0.9, mode="undirected"))
plot(graph.adjacency(mat>=0.9, mode="undirected"), layout=layout.circle)
dev.off()
install.packages("igraph")
library(igraph)
# 乱数から成る100×100のデータ行列を作成
mat <- matrix(runif(10000), nr=100)
png("110212_graph.png")
par(mfrow=c(2,1))
plot(graph.adjacency(mat>=0.9, mode="undirected"))
plot(graph.adjacency(mat>=0.9, mode="undirected"), layout=layout.circle)
dev.off()
Open Babelによる分子構造エネルギーの計算
Open bABERLという化学構造ファイル形式の変換を目的に開発されたソフトをまずはインストールする。
$ sudo apt-get install openbabel
$ obenergy CID_2244.sdf
A T O M T Y P E S
IDX TYPE
1 0800
2 0800
3 0801
4 0801
5 0603
6 0603
7 0603
8 0603
9 0603
10 0603
11 0601
12 0601
13 0600
14 0100
15 0100
16 0100
17 0100
18 0100
19 0100
20 0100
21 0100
C H A R G E S
IDX CHARGE
1 0.000000
2 -0.250000
3 -0.100000
4 -0.100000
5 0.000000
6 0.000000
7 -0.100000
8 -0.100000
9 -0.100000
10 -0.100000
11 0.100000
12 0.100000
13 0.000000
14 0.100000
15 0.100000
16 0.100000
17 0.100000
18 0.000000
19 0.000000
20 0.000000
21 0.250000
S E T T I N G U P C A L C U L A T I O N S
SETTING UP BOND CALCULATIONS...
SETTING UP ANGLE CALCULATIONS...
SETTING UP TORSION CALCULATIONS...
SETTING UP VAN DER WAALS CALCULATIONS...
SETTING UP ELECTROSTATIC CALCULATIONS...
E N E R G Y
TOTAL BOND STRETCHING ENERGY = 14203.861 kJ/mol
TOTAL ANGLE BENDING ENERGY = 1685.192 kJ/mol
TOTAL TORSIONAL ENERGY = 6.883 kJ/mol
TOTAL VAN DER WAALS ENERGY = 17385.054 kJ/mol
TOTAL ELECTROSTATIC ENERGY = -3.713 kJ/mol
TOTAL ENERGY = 33277.277 kJ/mol
==============================
*** Open Babel Warning in ReadMolecule
WARNING: Problems reading a MDL file
Cannot read creator/dimension line line
#こういう物理化学的な計算が瞬時にできるのがLinuxを使っているメリットのひとつやろうなーー♪
$ sudo apt-get install openbabel
$ obenergy CID_2244.sdf
A T O M T Y P E S
IDX TYPE
1 0800
2 0800
3 0801
4 0801
5 0603
6 0603
7 0603
8 0603
9 0603
10 0603
11 0601
12 0601
13 0600
14 0100
15 0100
16 0100
17 0100
18 0100
19 0100
20 0100
21 0100
C H A R G E S
IDX CHARGE
1 0.000000
2 -0.250000
3 -0.100000
4 -0.100000
5 0.000000
6 0.000000
7 -0.100000
8 -0.100000
9 -0.100000
10 -0.100000
11 0.100000
12 0.100000
13 0.000000
14 0.100000
15 0.100000
16 0.100000
17 0.100000
18 0.000000
19 0.000000
20 0.000000
21 0.250000
S E T T I N G U P C A L C U L A T I O N S
SETTING UP BOND CALCULATIONS...
SETTING UP ANGLE CALCULATIONS...
SETTING UP TORSION CALCULATIONS...
SETTING UP VAN DER WAALS CALCULATIONS...
SETTING UP ELECTROSTATIC CALCULATIONS...
E N E R G Y
TOTAL BOND STRETCHING ENERGY = 14203.861 kJ/mol
TOTAL ANGLE BENDING ENERGY = 1685.192 kJ/mol
TOTAL TORSIONAL ENERGY = 6.883 kJ/mol
TOTAL VAN DER WAALS ENERGY = 17385.054 kJ/mol
TOTAL ELECTROSTATIC ENERGY = -3.713 kJ/mol
TOTAL ENERGY = 33277.277 kJ/mol
==============================
*** Open Babel Warning in ReadMolecule
WARNING: Problems reading a MDL file
Cannot read creator/dimension line line
#こういう物理化学的な計算が瞬時にできるのがLinuxを使っているメリットのひとつやろうなーー♪
mol2psによりsdf->psの変換をしたどーーーー♪
pubchemにいって、アスピリンを入力して構造式のデータをとってくる。
このとき、
1 Download SDF
2 2D SDF save
の2段階で行った。
でも、以下の用にしてもできてしまうことを発見した
$ wget http://pubchem.ncbi.nlm.nih.gov/summary/summary.cgi?cid=2244
一般化すると
$ wget http://pubchem.ncbi.nlm.nih.gov/summary/summary.cgi?cid=xxxx
で好きな化合物のsdfファイルがとってこれそうだ!!!
それでダウンロードしたファイルをカレントディレクトリに移動しといて、
$ head CID_2244.sdf
2244
-OEChem-02121112262D
21 21 0 0 0 0 0 0 0999 V2000
3.7320 -0.0600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.3301 1.4400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 1.4400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 -1.5600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 -0.5600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4641 -0.0600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
ここで、mol2psという、MOLファイル表示をPostscriptに出力するソフトウェアをインストールする。
$ wget http://merian.pch.univie.ac.at/pch/download/chemistry\
> /mol2ps/bin/mol2ps-latest-linux-i386.gz
$ gunzip mol2ps-latest-linux-i386.gz
$ sudo install mol2ps-latest-linux-i386 /usr/local/bin/mol2ps
よし、実際ps形式に変更してみるぞ!!
$ mol2ps CID_2244.sdf > CID_2244.ps
$ head CID_2244.ps
%!PS-Adobe-2.0
%%Creator: mol2ps 0.2, Norbert Haider, University of Vienna, 2011
%%Title: CID_2244.sdf
% the following settings were used:
% font: Helvetica 14 pt (9 pt for subscripts)
% line width: 1.0
% automatic rotation:
% 0.00� around X axis
% 0.00� around Y axis
% 0.00� around Z axis
やはりブログにはpngファイルしか貼れないのでファイル形式を変換していく。
$ sudo apt-get install gv
$ ps2pdf CID_2244.ps
$ convert CID_2244.pdf -CID_2244.png
局所配列・大域配列
大域的配列
2つの配列の全体を比較し、たがいに類似している領域を可能なかぎり長く、そしてギャップをできるだけ短くするように整列させるもの。Neeedleman-Wunschのアルゴリズムが基本。Needleman-Wunschのアルゴリズムは、動的計画法によってあらかじめ与えられた一致・不一致、ギャップのスコアを元に整列配列のスコアを計算し、もっとも高いスコアになるように2本の配列をペアワイズで整列させるアルゴリズム。整列のすべての可能性を探索し、その中から最適な整列を得ることができる。
$ needle
Needleman-Wunsch global alignment of two sequences
Input sequence: refseqp:NP_203124
Second sequence(s): refseqp:NP_001018443
Gap opening penalty [10.0]: #Enter
Gap extension penalty [0.5]: #Enter
Output alignment [np_203124.needle]: #Enter
#生成したファイル"np_203124.needle"をのぞいてみる♪♪
$ cat np_203124.needle
########################################
# Program: needle
# Rundate: Sun 13 Feb 2011 01:05:46
# Commandline: needle
# -asequence refseqp:NP_203124
# -bsequence refseqp:NP_001018443
# Align_format: srspair
# Report_file: np_203124.needle
########################################
#=======================================
#
# Aligned_sequences: 2
# 1: NP_203124
# 2: NP_001018443
# Matrix: EBLOSUM62
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 345
# Identity: 204/345 (59.1%)
# Similarity: 241/345 (69.9%)
# Gaps: 38/345 (11.0%)
# Score: 1040.0
#
#
#=======================================
NP_203124 1 MDCVGWPPGRKWHLEKNTSCGGSSGICASYVTQMADDQGCIEEQGVEDS- 49
:|......:..:...:||. :||
NP_001018443 1 -------------------------MCRVDKEALTSENEVLEED--QDSY 23
NP_203124 50 ANEDSVDAKPDRSSFVPSLFSKKKKN---VTMRSIKTTRDRV--PTYQYN 94
..||..||||||.... .||...||| :..:....:..|: ||:||.
NP_001018443 24 GEEDVTDAKPDRKGRF-RLFGNFKKNDGKLQEKGESESHYRIVSPTFQYK 72
NP_203124 95 MNFEKLGKCIIINNKNFDKVTGMGVRNGTDKDAEALFKCFRSLGFDVIVY 144
|:.:::||||||||||||:.|||.||||||:||..|||||:||||||.||
NP_001018443 73 MSHQRVGKCIIINNKNFDEKTGMNVRNGTDRDAGELFKCFKSLGFDVAVY 122
NP_203124 145 NDCSCAKMQDLLKKASEEDHTNAACFACILLSHGEENVIYGKDGVTPIKD 194
||.:|..|:.|||..|||||::::||||||||||||.:|||.||..|||.
NP_001018443 123 NDQTCRNMERLLKAVSEEDHSDSSCFACILLSHGEEGMIYGTDGAMPIKT 172
NP_203124 195 LTAHFRGDRCKTLLEKPKLFFIQACRGTELDDGIQADSGPIND---TDAN 241
:|:.|:||.||:|:.||||||||||||:|.|||:|.||||.|| ||||
NP_001018443 173 MTSLFKGDVCKSLVGKPKLFFIQACRGSEFDDGVQTDSGPPNDTIETDAN 222
NP_203124 242 PRYKIPVEADFLFAYSTVPGYYSWRSPGRGSWFVQALCSILEEHGKDLEI 291
||:||||||||||||||||||||||:||||||||||||::|.|.||.|||
NP_001018443 223 PRHKIPVEADFLFAYSTVPGYYSWRNPGRGSWFVQALCNVLSEFGKQLEI 272
NP_203124 292 MQILTRVNDRVARHFESQSDDPHFHEKKQIPCVVSMLTKELYFSQ 336
||||||||..||..|||.|:||.|.||||||||||||||||||:
NP_001018443 273 MQILTRVNYMVATSFESWSEDPRFSEKKQIPCVVSMLTKELYFN- 316
#---------------------------------------
#---------------------------------------
次に、局所的整列をさせてみる。
局所的整列とは、配列の局所的に類似している領域を探し、整列させるもの、局所整列をさせるためにはSmith-Watermanのアルゴリズムが基本になる。これは上のNeedleman-Wunschのアルゴリズムの特殊な形である。Smith-Watermanのアルゴリズムでは局所的な類似性を見て整列させたいので、スコアがマイナスになるとその時点でスコアを0にしてしまう。Needleman-Wunschのアルゴリズムではは率の端から端までを整列させたが、Smith-Watermanのアルゴリズムでは、もっともスコアが高い部分から整列させる。
$ water
Smith-Waterman local alignment of sequences
Input sequence: refseqp:NP_203124
Second sequence(s): refseqp:NP_001018443
Gap opening penalty [10.0]: #Enter
Gap extension penalty [0.5]: #Enter
Output alignment [np_203124.water]: #Enter
#できたファイルをのぞいてみる。
$ cat np_203124.water
########################################
# Program: needle
# Rundate: Sun 13 Feb 2011 01:05:46
# Commandline: needle
# -asequence refseqp:NP_203124
# -bsequence refseqp:NP_001018443
# Align_format: srspair
# Report_file: np_203124.needle
########################################
#=======================================
#
# Aligned_sequences: 2
# 1: NP_203124
# 2: NP_001018443
# Matrix: EBLOSUM62
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 345
# Identity: 204/345 (59.1%)
# Similarity: 241/345 (69.9%)
# Gaps: 38/345 (11.0%)
# Score: 1040.0
#
#
#=======================================
NP_203124 1 MDCVGWPPGRKWHLEKNTSCGGSSGICASYVTQMADDQGCIEEQGVEDS- 49
:|......:..:...:||. :||
NP_001018443 1 -------------------------MCRVDKEALTSENEVLEED--QDSY 23
NP_203124 50 ANEDSVDAKPDRSSFVPSLFSKKKKN---VTMRSIKTTRDRV--PTYQYN 94
..||..||||||.... .||...||| :..:....:..|: ||:||.
NP_001018443 24 GEEDVTDAKPDRKGRF-RLFGNFKKNDGKLQEKGESESHYRIVSPTFQYK 72
NP_203124 95 MNFEKLGKCIIINNKNFDKVTGMGVRNGTDKDAEALFKCFRSLGFDVIVY 144
|:.:::||||||||||||:.|||.||||||:||..|||||:||||||.||
NP_001018443 73 MSHQRVGKCIIINNKNFDEKTGMNVRNGTDRDAGELFKCFKSLGFDVAVY 122
NP_203124 145 NDCSCAKMQDLLKKASEEDHTNAACFACILLSHGEENVIYGKDGVTPIKD 194
||.:|..|:.|||..|||||::::||||||||||||.:|||.||..|||.
NP_001018443 123 NDQTCRNMERLLKAVSEEDHSDSSCFACILLSHGEEGMIYGTDGAMPIKT 172
NP_203124 195 LTAHFRGDRCKTLLEKPKLFFIQACRGTELDDGIQADSGPIND---TDAN 241
:|:.|:||.||:|:.||||||||||||:|.|||:|.||||.|| ||||
NP_001018443 173 MTSLFKGDVCKSLVGKPKLFFIQACRGSEFDDGVQTDSGPPNDTIETDAN 222
NP_203124 242 PRYKIPVEADFLFAYSTVPGYYSWRSPGRGSWFVQALCSILEEHGKDLEI 291
||:||||||||||||||||||||||:||||||||||||::|.|.||.|||
NP_001018443 223 PRHKIPVEADFLFAYSTVPGYYSWRNPGRGSWFVQALCNVLSEFGKQLEI 272
NP_203124 292 MQILTRVNDRVARHFESQSDDPHFHEKKQIPCVVSMLTKELYFSQ 336
||||||||..||..|||.|:||.|.||||||||||||||||||:
NP_001018443 273 MQILTRVNYMVATSFESWSEDPRFSEKKQIPCVVSMLTKELYFN- 316
#---------------------------------------
#---------------------------------------
今回のアライメントでは結果が一緒であったが、一般的に、waterでやると、無理して配列しないため、アライメントの対処から外れてしむ配列が生じがちである。
今回は、vertebrate同士の比較的近いアミノ酸配列に対してのアライメントだったからよかったけどね。
2つの配列の全体を比較し、たがいに類似している領域を可能なかぎり長く、そしてギャップをできるだけ短くするように整列させるもの。Neeedleman-Wunschのアルゴリズムが基本。Needleman-Wunschのアルゴリズムは、動的計画法によってあらかじめ与えられた一致・不一致、ギャップのスコアを元に整列配列のスコアを計算し、もっとも高いスコアになるように2本の配列をペアワイズで整列させるアルゴリズム。整列のすべての可能性を探索し、その中から最適な整列を得ることができる。
$ needle
Needleman-Wunsch global alignment of two sequences
Input sequence: refseqp:NP_203124
Second sequence(s): refseqp:NP_001018443
Gap opening penalty [10.0]: #Enter
Gap extension penalty [0.5]: #Enter
Output alignment [np_203124.needle]: #Enter
#生成したファイル"np_203124.needle"をのぞいてみる♪♪
$ cat np_203124.needle
########################################
# Program: needle
# Rundate: Sun 13 Feb 2011 01:05:46
# Commandline: needle
# -asequence refseqp:NP_203124
# -bsequence refseqp:NP_001018443
# Align_format: srspair
# Report_file: np_203124.needle
########################################
#=======================================
#
# Aligned_sequences: 2
# 1: NP_203124
# 2: NP_001018443
# Matrix: EBLOSUM62
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 345
# Identity: 204/345 (59.1%)
# Similarity: 241/345 (69.9%)
# Gaps: 38/345 (11.0%)
# Score: 1040.0
#
#
#=======================================
NP_203124 1 MDCVGWPPGRKWHLEKNTSCGGSSGICASYVTQMADDQGCIEEQGVEDS- 49
:|......:..:...:||. :||
NP_001018443 1 -------------------------MCRVDKEALTSENEVLEED--QDSY 23
NP_203124 50 ANEDSVDAKPDRSSFVPSLFSKKKKN---VTMRSIKTTRDRV--PTYQYN 94
..||..||||||.... .||...||| :..:....:..|: ||:||.
NP_001018443 24 GEEDVTDAKPDRKGRF-RLFGNFKKNDGKLQEKGESESHYRIVSPTFQYK 72
NP_203124 95 MNFEKLGKCIIINNKNFDKVTGMGVRNGTDKDAEALFKCFRSLGFDVIVY 144
|:.:::||||||||||||:.|||.||||||:||..|||||:||||||.||
NP_001018443 73 MSHQRVGKCIIINNKNFDEKTGMNVRNGTDRDAGELFKCFKSLGFDVAVY 122
NP_203124 145 NDCSCAKMQDLLKKASEEDHTNAACFACILLSHGEENVIYGKDGVTPIKD 194
||.:|..|:.|||..|||||::::||||||||||||.:|||.||..|||.
NP_001018443 123 NDQTCRNMERLLKAVSEEDHSDSSCFACILLSHGEEGMIYGTDGAMPIKT 172
NP_203124 195 LTAHFRGDRCKTLLEKPKLFFIQACRGTELDDGIQADSGPIND---TDAN 241
:|:.|:||.||:|:.||||||||||||:|.|||:|.||||.|| ||||
NP_001018443 173 MTSLFKGDVCKSLVGKPKLFFIQACRGSEFDDGVQTDSGPPNDTIETDAN 222
NP_203124 242 PRYKIPVEADFLFAYSTVPGYYSWRSPGRGSWFVQALCSILEEHGKDLEI 291
||:||||||||||||||||||||||:||||||||||||::|.|.||.|||
NP_001018443 223 PRHKIPVEADFLFAYSTVPGYYSWRNPGRGSWFVQALCNVLSEFGKQLEI 272
NP_203124 292 MQILTRVNDRVARHFESQSDDPHFHEKKQIPCVVSMLTKELYFSQ 336
||||||||..||..|||.|:||.|.||||||||||||||||||:
NP_001018443 273 MQILTRVNYMVATSFESWSEDPRFSEKKQIPCVVSMLTKELYFN- 316
#---------------------------------------
#---------------------------------------
次に、局所的整列をさせてみる。
局所的整列とは、配列の局所的に類似している領域を探し、整列させるもの、局所整列をさせるためにはSmith-Watermanのアルゴリズムが基本になる。これは上のNeedleman-Wunschのアルゴリズムの特殊な形である。Smith-Watermanのアルゴリズムでは局所的な類似性を見て整列させたいので、スコアがマイナスになるとその時点でスコアを0にしてしまう。Needleman-Wunschのアルゴリズムではは率の端から端までを整列させたが、Smith-Watermanのアルゴリズムでは、もっともスコアが高い部分から整列させる。
$ water
Smith-Waterman local alignment of sequences
Input sequence: refseqp:NP_203124
Second sequence(s): refseqp:NP_001018443
Gap opening penalty [10.0]: #Enter
Gap extension penalty [0.5]: #Enter
Output alignment [np_203124.water]: #Enter
#できたファイルをのぞいてみる。
$ cat np_203124.water
########################################
# Program: needle
# Rundate: Sun 13 Feb 2011 01:05:46
# Commandline: needle
# -asequence refseqp:NP_203124
# -bsequence refseqp:NP_001018443
# Align_format: srspair
# Report_file: np_203124.needle
########################################
#=======================================
#
# Aligned_sequences: 2
# 1: NP_203124
# 2: NP_001018443
# Matrix: EBLOSUM62
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 345
# Identity: 204/345 (59.1%)
# Similarity: 241/345 (69.9%)
# Gaps: 38/345 (11.0%)
# Score: 1040.0
#
#
#=======================================
NP_203124 1 MDCVGWPPGRKWHLEKNTSCGGSSGICASYVTQMADDQGCIEEQGVEDS- 49
:|......:..:...:||. :||
NP_001018443 1 -------------------------MCRVDKEALTSENEVLEED--QDSY 23
NP_203124 50 ANEDSVDAKPDRSSFVPSLFSKKKKN---VTMRSIKTTRDRV--PTYQYN 94
..||..||||||.... .||...||| :..:....:..|: ||:||.
NP_001018443 24 GEEDVTDAKPDRKGRF-RLFGNFKKNDGKLQEKGESESHYRIVSPTFQYK 72
NP_203124 95 MNFEKLGKCIIINNKNFDKVTGMGVRNGTDKDAEALFKCFRSLGFDVIVY 144
|:.:::||||||||||||:.|||.||||||:||..|||||:||||||.||
NP_001018443 73 MSHQRVGKCIIINNKNFDEKTGMNVRNGTDRDAGELFKCFKSLGFDVAVY 122
NP_203124 145 NDCSCAKMQDLLKKASEEDHTNAACFACILLSHGEENVIYGKDGVTPIKD 194
||.:|..|:.|||..|||||::::||||||||||||.:|||.||..|||.
NP_001018443 123 NDQTCRNMERLLKAVSEEDHSDSSCFACILLSHGEEGMIYGTDGAMPIKT 172
NP_203124 195 LTAHFRGDRCKTLLEKPKLFFIQACRGTELDDGIQADSGPIND---TDAN 241
:|:.|:||.||:|:.||||||||||||:|.|||:|.||||.|| ||||
NP_001018443 173 MTSLFKGDVCKSLVGKPKLFFIQACRGSEFDDGVQTDSGPPNDTIETDAN 222
NP_203124 242 PRYKIPVEADFLFAYSTVPGYYSWRSPGRGSWFVQALCSILEEHGKDLEI 291
||:||||||||||||||||||||||:||||||||||||::|.|.||.|||
NP_001018443 223 PRHKIPVEADFLFAYSTVPGYYSWRNPGRGSWFVQALCNVLSEFGKQLEI 272
NP_203124 292 MQILTRVNDRVARHFESQSDDPHFHEKKQIPCVVSMLTKELYFSQ 336
||||||||..||..|||.|:||.|.||||||||||||||||||:
NP_001018443 273 MQILTRVNYMVATSFESWSEDPRFSEKKQIPCVVSMLTKELYFN- 316
#---------------------------------------
#---------------------------------------
今回のアライメントでは結果が一緒であったが、一般的に、waterでやると、無理して配列しないため、アライメントの対処から外れてしむ配列が生じがちである。
今回は、vertebrate同士の比較的近いアミノ酸配列に対してのアライメントだったからよかったけどね。
必見!BioRubyのUbuntuへのインストール方法。
RubyはデフォルトでUbuntuに入っているけど、BioRubyは入っていない。その上、apt-get installコマンドではインストールできひんかった。そこで、ソースコードからインストールを試みてみた。
$ wget http://bioruby.open-bio.org/archive/bioruby-1.4.0
#これで、http://bioruby.open-bio.org/archiveにおいてある、bioruby-1.4.0というパッケージがとってこれるぜい♪CUIでやるなら以下にプリントスクリーンを貼っておきました☆
$ cd bioruby-1.4.0
$ tar xfz bioruby-1.4.0 #これで解答できてワンサカ、カレントディレクトリにファイルが生成するはず♪
$ pwd
/home/kappa/2011/1102/bioruby-1.4.0
$ ./configure
bash: ./configure: No such file or directory
#ウギャ!なんかようわからんことになった。
#ググッた結果以下。
ーーーーーーここからーーーーーー
bash: ./configure: No such file or directory
この意味は「./」の場所に「configure」なんてファイルもディレクトリも無いですよという意味です。「./」は通常自分がいるディレクトリの場所を表しています。
今自分がいるディレクトリに(通常ソースを解凍して出来たディレクトリ)にconfigureというファイルは無いでしょうか?
無いならばhir0さんがおっしゃっているように最初からconfigureファイルがない場合もあります。
その場合は大抵ソースを解凍した中にINSTALL、README、TODOなどのファイルがあるのでまずそれを読んでみましょう。
また解凍した中にbootstrap、bootstrap.sh、autogen.sh等のファイルがありconfigure(configure.inやconfigure.acではなく純粋にただのconfigureです)が
見当たらない場合はまず最初にbootstrapやautogen.shを実行させてconfigureファイルを自動作成する必要があります。
ーーーーーここまでーーーーーー
#なるほど。全部、configure入力しまくればいいわけではないわけね。よく勉強になりました。
#README読んでちょっと頑張ってみた。
$ ruby setup.rb
$ sudo ruby setup.rb
$ ruby setup.rb config
$ ruby setup.rb install
#これでインストールできた!!・・・・はず。。。
#テスト用のコマンドもちゃんとありました♪
$ ruby setup.rb test
Running tests...
Loaded suite test
Started
....................................................................F............F...............................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................FF.................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................
Finished in 450.006201 seconds.
1) Failure:
test_esearch_retmax_retstart(Bio::FuncTestPubmedClassMethod) [./test/functional/bio/io/test_pubmed.rb:57]:
The failure may be caused by changes of NCBI PubMed.
<35> expected but was
<20>.
2) Failure:
test_retrieve_1id_1db(Bio::FuncTestTogoWSREST) [./test/functional/bio/io/test_togows.rb:142]:
<false> is not true.
3) Failure:
test_libxml(Bio::TestPhyloXMLWriter_Check_LibXML) [./test/unit/bio/db/test_phyloxml_writer.rb:31]:
Error: libxml-ruby library is not present. Please install libxml-ruby library. It is needed for Bio::PhyloXML module. Unit test for PhyloXML will not be performed.
<nil> is not true.
4) Failure:
test_libxml(Bio::TestPhyloXML_Check_LibXML) [./test/unit/bio/db/test_phyloxml.rb:29]:
Error: libxml-ruby library is not present. Please install libxml-ruby library. It is needed for Bio::PhyloXML module. Unit test for PhyloXML will not be performed.
<nil> is not true.
2564 tests, 19598 assertions, 4 failures, 0 errors
#errorは一つもなく、failureが4個出たようだ。これらに対してどう対処すればいいのか現段階ではよくわからない。
#とりあえず、気をとりなおして、BioRubyシェルを開いてみる♪♪♪
$ bioruby
Creating directory (/home/kappa/.bioruby/shell/session) ... done
Creating directory (/home/kappa/.bioruby/shell/plugin) ... done
Creating directory (/home/kappa/.bioruby/data) ... done
. . . B i o R u b y i n t h e s h e l l . . .
Version : BioRuby 1.4.0 / Ruby 1.8.7
bioruby> exit
. . . B i o R u b y i n t h e s h e l l . . .
Saving history (/home/kappa/.bioruby/shell/session/history) ... done
Saving object (/home/kappa/.bioruby/shell/session/object) ... done
Saving config (/home/kappa/.bioruby/shell/session/config) ... done
#なにやら、わんさかファイルが保存されてしまっているようだ。これは解決しなければならない。biorubyを立ち上げて、閉じるつどいらないファイルが増えたんじゃかなわない笑
ちなみに、BioRubyシェルでは、BioRubyライブラリの全機能をインタラクティブに利用できるだけでなく、BioRubyの中でよく使う機能をより短いコマンドとして書くこともできるようになっている。
$ wget http://bioruby.open-bio.org/archive/bioruby-1.4.0
#これで、http://bioruby.open-bio.org/archiveにおいてある、bioruby-1.4.0というパッケージがとってこれるぜい♪CUIでやるなら以下にプリントスクリーンを貼っておきました☆
$ cd bioruby-1.4.0
$ tar xfz bioruby-1.4.0 #これで解答できてワンサカ、カレントディレクトリにファイルが生成するはず♪
$ pwd
/home/kappa/2011/1102/bioruby-1.4.0
$ ./configure
bash: ./configure: No such file or directory
#ウギャ!なんかようわからんことになった。
#ググッた結果以下。
ーーーーーーここからーーーーーー
bash: ./configure: No such file or directory
この意味は「./」の場所に「configure」なんてファイルもディレクトリも無いですよという意味です。「./」は通常自分がいるディレクトリの場所を表しています。
今自分がいるディレクトリに(通常ソースを解凍して出来たディレクトリ)にconfigureというファイルは無いでしょうか?
無いならばhir0さんがおっしゃっているように最初からconfigureファイルがない場合もあります。
その場合は大抵ソースを解凍した中にINSTALL、README、TODOなどのファイルがあるのでまずそれを読んでみましょう。
また解凍した中にbootstrap、bootstrap.sh、autogen.sh等のファイルがありconfigure(configure.inやconfigure.acではなく純粋にただのconfigureです)が
見当たらない場合はまず最初にbootstrapやautogen.shを実行させてconfigureファイルを自動作成する必要があります。
ーーーーーここまでーーーーーー
#なるほど。全部、configure入力しまくればいいわけではないわけね。よく勉強になりました。
#README読んでちょっと頑張ってみた。
$ ruby setup.rb
$ sudo ruby setup.rb
$ ruby setup.rb config
$ ruby setup.rb install
#これでインストールできた!!・・・・はず。。。
#テスト用のコマンドもちゃんとありました♪
$ ruby setup.rb test
Running tests...
Loaded suite test
Started
....................................................................F............F...............................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................FF.................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................................
Finished in 450.006201 seconds.
1) Failure:
test_esearch_retmax_retstart(Bio::FuncTestPubmedClassMethod) [./test/functional/bio/io/test_pubmed.rb:57]:
The failure may be caused by changes of NCBI PubMed.
<35> expected but was
<20>.
2) Failure:
test_retrieve_1id_1db(Bio::FuncTestTogoWSREST) [./test/functional/bio/io/test_togows.rb:142]:
<false> is not true.
3) Failure:
test_libxml(Bio::TestPhyloXMLWriter_Check_LibXML) [./test/unit/bio/db/test_phyloxml_writer.rb:31]:
Error: libxml-ruby library is not present. Please install libxml-ruby library. It is needed for Bio::PhyloXML module. Unit test for PhyloXML will not be performed.
<nil> is not true.
4) Failure:
test_libxml(Bio::TestPhyloXML_Check_LibXML) [./test/unit/bio/db/test_phyloxml.rb:29]:
Error: libxml-ruby library is not present. Please install libxml-ruby library. It is needed for Bio::PhyloXML module. Unit test for PhyloXML will not be performed.
<nil> is not true.
2564 tests, 19598 assertions, 4 failures, 0 errors
#errorは一つもなく、failureが4個出たようだ。これらに対してどう対処すればいいのか現段階ではよくわからない。
#とりあえず、気をとりなおして、BioRubyシェルを開いてみる♪♪♪
$ bioruby
Creating directory (/home/kappa/.bioruby/shell/session) ... done
Creating directory (/home/kappa/.bioruby/shell/plugin) ... done
Creating directory (/home/kappa/.bioruby/data) ... done
. . . B i o R u b y i n t h e s h e l l . . .
Version : BioRuby 1.4.0 / Ruby 1.8.7
bioruby> exit
. . . B i o R u b y i n t h e s h e l l . . .
Saving history (/home/kappa/.bioruby/shell/session/history) ... done
Saving object (/home/kappa/.bioruby/shell/session/object) ... done
Saving config (/home/kappa/.bioruby/shell/session/config) ... done
#なにやら、わんさかファイルが保存されてしまっているようだ。これは解決しなければならない。biorubyを立ち上げて、閉じるつどいらないファイルが増えたんじゃかなわない笑
ちなみに、BioRubyシェルでは、BioRubyライブラリの全機能をインタラクティブに利用できるだけでなく、BioRubyの中でよく使う機能をより短いコマンドとして書くこともできるようになっている。
BLASTの置換行列を取ってきた☆
二つの配列があるとして、それらをアライメントするときは、あらかじめスコアリング方法を決めておく必要がある。これに用いられるのが置換行列である。アミノ酸や核酸の置換のしやすさを表した行列で、さまざまな置換行列が提案されている。アミノ酸の置換行列としては、BLOSUMが有名である。BLOCKSデータベース中のたんぱく質ファミリーでよく保存されている領域でアミノ酸の相対頻度と置換確率を測定し20種類のアミノ酸について全ペア間の置換それぞれについて対数オッズ比を算出したものである。BLOSUM62は、BLASTのデフォルトの置換行列であり、62%以上の配列一致性を示すアミノ酸配列間で観測された置換を基に算出して得られている。
$ wget ftp://ftp.ncbi.nih.gov/blast/matrices/BLOSUM62
$ cat BLOSUM62
# Matrix made by matblas from blosum62.iij
# * column uses minimum score
# BLOSUM Clustered Scoring Matrix in 1/2 Bit Units
# Blocks Database = /data/blocks_5.0/blocks.dat
# Cluster Percentage: >= 62
# Entropy = 0.6979, Expected = -0.5209
A R N D C Q E G H I L K M F P S T W Y V B Z X *
A 4 -1 -2 -2 0 -1 -1 0 -2 -1 -1 -1 -1 -2 -1 1 0 -3 -2 0 -2 -1 0 -4
R -1 5 0 -2 -3 1 0 -2 0 -3 -2 2 -1 -3 -2 -1 -1 -3 -2 -3 -1 0 -1 -4
N -2 0 6 1 -3 0 0 0 1 -3 -3 0 -2 -3 -2 1 0 -4 -2 -3 3 0 -1 -4
D -2 -2 1 6 -3 0 2 -1 -1 -3 -4 -1 -3 -3 -1 0 -1 -4 -3 -3 4 1 -1 -4
C 0 -3 -3 -3 9 -3 -4 -3 -3 -1 -1 -3 -1 -2 -3 -1 -1 -2 -2 -1 -3 -3 -2 -4
Q -1 1 0 0 -3 5 2 -2 0 -3 -2 1 0 -3 -1 0 -1 -2 -1 -2 0 3 -1 -4
E -1 0 0 2 -4 2 5 -2 0 -3 -3 1 -2 -3 -1 0 -1 -3 -2 -2 1 4 -1 -4
G 0 -2 0 -1 -3 -2 -2 6 -2 -4 -4 -2 -3 -3 -2 0 -2 -2 -3 -3 -1 -2 -1 -4
H -2 0 1 -1 -3 0 0 -2 8 -3 -3 -1 -2 -1 -2 -1 -2 -2 2 -3 0 0 -1 -4
I -1 -3 -3 -3 -1 -3 -3 -4 -3 4 2 -3 1 0 -3 -2 -1 -3 -1 3 -3 -3 -1 -4
L -1 -2 -3 -4 -1 -2 -3 -4 -3 2 4 -2 2 0 -3 -2 -1 -2 -1 1 -4 -3 -1 -4
K -1 2 0 -1 -3 1 1 -2 -1 -3 -2 5 -1 -3 -1 0 -1 -3 -2 -2 0 1 -1 -4
M -1 -1 -2 -3 -1 0 -2 -3 -2 1 2 -1 5 0 -2 -1 -1 -1 -1 1 -3 -1 -1 -4
F -2 -3 -3 -3 -2 -3 -3 -3 -1 0 0 -3 0 6 -4 -2 -2 1 3 -1 -3 -3 -1 -4
P -1 -2 -2 -1 -3 -1 -1 -2 -2 -3 -3 -1 -2 -4 7 -1 -1 -4 -3 -2 -2 -1 -2 -4
S 1 -1 1 0 -1 0 0 0 -1 -2 -2 0 -1 -2 -1 4 1 -3 -2 -2 0 0 0 -4
T 0 -1 0 -1 -1 -1 -1 -2 -2 -1 -1 -1 -1 -2 -1 1 5 -2 -2 0 -1 -1 0 -4
W -3 -3 -4 -4 -2 -2 -3 -2 -2 -3 -2 -3 -1 1 -4 -3 -2 11 2 -3 -4 -3 -2 -4
Y -2 -2 -2 -3 -2 -1 -2 -3 2 -1 -1 -2 -1 3 -3 -2 -2 2 7 -1 -3 -2 -1 -4
V 0 -3 -3 -3 -1 -2 -2 -3 -3 3 1 -2 1 -1 -2 -2 0 -3 -1 4 -3 -2 -1 -4
B -2 -1 3 4 -3 0 1 -1 0 -3 -4 0 -3 -3 -2 0 -1 -4 -3 -3 4 1 -1 -4
Z -1 0 0 1 -3 3 4 -2 0 -3 -3 1 -1 -3 -1 0 -1 -3 -2 -2 1 4 -1 -4
X 0 -1 -1 -1 -2 -1 -1 -1 -1 -1 -1 -1 -1 -1 -2 0 0 -2 -1 -1 -1 -1 -1 -4
* -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 1
$ wget ftp://ftp.ncbi.nih.gov/blast/matrices/BLOSUM62
$ cat BLOSUM62
# Matrix made by matblas from blosum62.iij
# * column uses minimum score
# BLOSUM Clustered Scoring Matrix in 1/2 Bit Units
# Blocks Database = /data/blocks_5.0/blocks.dat
# Cluster Percentage: >= 62
# Entropy = 0.6979, Expected = -0.5209
A R N D C Q E G H I L K M F P S T W Y V B Z X *
A 4 -1 -2 -2 0 -1 -1 0 -2 -1 -1 -1 -1 -2 -1 1 0 -3 -2 0 -2 -1 0 -4
R -1 5 0 -2 -3 1 0 -2 0 -3 -2 2 -1 -3 -2 -1 -1 -3 -2 -3 -1 0 -1 -4
N -2 0 6 1 -3 0 0 0 1 -3 -3 0 -2 -3 -2 1 0 -4 -2 -3 3 0 -1 -4
D -2 -2 1 6 -3 0 2 -1 -1 -3 -4 -1 -3 -3 -1 0 -1 -4 -3 -3 4 1 -1 -4
C 0 -3 -3 -3 9 -3 -4 -3 -3 -1 -1 -3 -1 -2 -3 -1 -1 -2 -2 -1 -3 -3 -2 -4
Q -1 1 0 0 -3 5 2 -2 0 -3 -2 1 0 -3 -1 0 -1 -2 -1 -2 0 3 -1 -4
E -1 0 0 2 -4 2 5 -2 0 -3 -3 1 -2 -3 -1 0 -1 -3 -2 -2 1 4 -1 -4
G 0 -2 0 -1 -3 -2 -2 6 -2 -4 -4 -2 -3 -3 -2 0 -2 -2 -3 -3 -1 -2 -1 -4
H -2 0 1 -1 -3 0 0 -2 8 -3 -3 -1 -2 -1 -2 -1 -2 -2 2 -3 0 0 -1 -4
I -1 -3 -3 -3 -1 -3 -3 -4 -3 4 2 -3 1 0 -3 -2 -1 -3 -1 3 -3 -3 -1 -4
L -1 -2 -3 -4 -1 -2 -3 -4 -3 2 4 -2 2 0 -3 -2 -1 -2 -1 1 -4 -3 -1 -4
K -1 2 0 -1 -3 1 1 -2 -1 -3 -2 5 -1 -3 -1 0 -1 -3 -2 -2 0 1 -1 -4
M -1 -1 -2 -3 -1 0 -2 -3 -2 1 2 -1 5 0 -2 -1 -1 -1 -1 1 -3 -1 -1 -4
F -2 -3 -3 -3 -2 -3 -3 -3 -1 0 0 -3 0 6 -4 -2 -2 1 3 -1 -3 -3 -1 -4
P -1 -2 -2 -1 -3 -1 -1 -2 -2 -3 -3 -1 -2 -4 7 -1 -1 -4 -3 -2 -2 -1 -2 -4
S 1 -1 1 0 -1 0 0 0 -1 -2 -2 0 -1 -2 -1 4 1 -3 -2 -2 0 0 0 -4
T 0 -1 0 -1 -1 -1 -1 -2 -2 -1 -1 -1 -1 -2 -1 1 5 -2 -2 0 -1 -1 0 -4
W -3 -3 -4 -4 -2 -2 -3 -2 -2 -3 -2 -3 -1 1 -4 -3 -2 11 2 -3 -4 -3 -2 -4
Y -2 -2 -2 -3 -2 -1 -2 -3 2 -1 -1 -2 -1 3 -3 -2 -2 2 7 -1 -3 -2 -1 -4
V 0 -3 -3 -3 -1 -2 -2 -3 -3 3 1 -2 1 -1 -2 -2 0 -3 -1 4 -3 -2 -1 -4
B -2 -1 3 4 -3 0 1 -1 0 -3 -4 0 -3 -3 -2 0 -1 -4 -3 -3 4 1 -1 -4
Z -1 0 0 1 -3 3 4 -2 0 -3 -3 1 -1 -3 -1 0 -1 -3 -2 -2 1 4 -1 -4
X 0 -1 -1 -1 -2 -1 -1 -1 -1 -1 -1 -1 -1 -1 -2 0 0 -2 -1 -1 -1 -1 -1 -4
* -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 -4 1
二つのアミノ酸配列に対してドットプロットを描写する。ps,pdf,tifの変換も勉強したよ♪
caspase7のヒトとzebrafishのホモログのペアワイズ作成においてドットプロットを描写する。両方の配列が同じアミノ酸や延期のときにに点が打たれる。
$ dottup
Displays a wordmatch dotplot of two sequences
Input sequence: refseqp:NP_203124 #ひとのCASP7
Second sequence: refseqp:NP_001018443 #ゼブラフィッシュのcasp7
Word size [10]: #ここはEnter
Graph type [x11]: ps
Created dottup.ps #psファイル作成をしている。
$ ls
110208.txt 110211.pl 110211.txt 110211_2.pl dottup.ps var0.pl var1.pl
$ sudo apt-get install gv
$ gv dottup.ps
$ ps2pdf dottup.ps #PostScriptファイルは利便性が低いのでpdfファイルに変更する。
$ ls
110208.txt 110211.pl 110211.txt 110211_2.pl dottup.pdf dottup.ps var0.pl var1.pl
$ sudo apt-get install xpdf-reader
$ xpdf dottup.pdf
$ convert dottup.pdf -dottup.tiff #さらにこっから、pdf -> tiff 変換を行う!!!!
$ convert dottup.pdf -dottup.png #pngファイルじゃないとブログに貼れなかったんで。。。
$ dottup
Displays a wordmatch dotplot of two sequences
Input sequence: refseqp:NP_203124 #ひとのCASP7
Second sequence: refseqp:NP_001018443 #ゼブラフィッシュのcasp7
Word size [10]: #ここはEnter
Graph type [x11]: ps
Created dottup.ps #psファイル作成をしている。
$ ls
110208.txt 110211.pl 110211.txt 110211_2.pl dottup.ps var0.pl var1.pl
$ sudo apt-get install gv
$ gv dottup.ps
$ ps2pdf dottup.ps #PostScriptファイルは利便性が低いのでpdfファイルに変更する。
$ ls
110208.txt 110211.pl 110211.txt 110211_2.pl dottup.pdf dottup.ps var0.pl var1.pl
$ sudo apt-get install xpdf-reader
$ xpdf dottup.pdf
$ convert dottup.pdf -dottup.tiff #さらにこっから、pdf -> tiff 変換を行う!!!!
$ convert dottup.pdf -dottup.png #pngファイルじゃないとブログに貼れなかったんで。。。
perlで変数2
$ vim 110211.pl
a
my $name = "programming Lesson";
print "\$name is $name\n";
my $name = "Programming" . "Lesson";
print "\$name is $name\n";
my $name = "Programming ". "Lesson";
print "\$name is $name\n";
my $name1 = "Programming";
my $name2 = "Lesson";
my $name = "$name1 $name2";
print "\$name is $name\n";
Esc
:wq
$ perl -cw 110211.pl
110211.pl syntax OK
$ perl 110211.pl #作ったプログラム110211.plの実行
$name is programming Lesson
$name is ProgrammingLesson
$name is Programming Lesson
$name is Programming Lesson
$ vim 110211_2.pl
a
use strict;
use warnings;
my $x ="123";
my $y ="456";
print $x + $y
print $x . $y;
Esc
perl -cw 110211_2.pl
1110211_2.pl syntax OK
$ perl 110211_2.pl
579
a
my $name = "programming Lesson";
print "\$name is $name\n";
my $name = "Programming" . "Lesson";
print "\$name is $name\n";
my $name = "Programming ". "Lesson";
print "\$name is $name\n";
my $name1 = "Programming";
my $name2 = "Lesson";
my $name = "$name1 $name2";
print "\$name is $name\n";
Esc
:wq
$ perl -cw 110211.pl
110211.pl syntax OK
$ perl 110211.pl #作ったプログラム110211.plの実行
$name is programming Lesson
$name is ProgrammingLesson
$name is Programming Lesson
$name is Programming Lesson
$ vim 110211_2.pl
a
use strict;
use warnings;
my $x ="123";
my $y ="456";
print $x + $y
print $x . $y;
Esc
perl -cw 110211_2.pl
1110211_2.pl syntax OK
$ perl 110211_2.pl
579
maSigProパッケージによる時系列データの処理方法
source("http://bioconductor.org/biocLite.R")
biocLite("maSigPro")
library(maSigPro)
data(edesign.abiotic)
data(data.abiotic)
edesign.abiotic
Time Replicate Control Cold Heat Salt
Control_3H_1 3 1 1 0 0 0
Control_3H_2 3 1 1 0 0 0
Control_3H_3 3 1 1 0 0 0
Control_9H_1 9 2 1 0 0 0
Control_9H_2 9 2 1 0 0 0
Control_9H_3 9 2 1 0 0 0
Control_27H_1 27 3 1 0 0 0
Control_27H_2 27 3 1 0 0 0
Control_27H_3 27 3 1 0 0 0
Cold_3H_1 3 4 0 1 0 0
Cold_3H_2 3 4 0 1 0 0
Cold_3H_3 3 4 0 1 0 0
Cold_9H_1 9 5 0 1 0 0
Cold_9H_2 9 5 0 1 0 0
Cold_9H_3 9 5 0 1 0 0
Cold_27H_1 27 6 0 1 0 0
Cold_27H_2 27 6 0 1 0 0
Cold_27H_3 27 6 0 1 0 0
Heat_3H_1 3 7 0 0 1 0
Heat_3H_2 3 7 0 0 1 0
Heat_3H_3 3 7 0 0 1 0
Heat_9H_1 9 8 0 0 1 0
Heat_9H_2 9 8 0 0 1 0
Heat_9H_3 9 8 0 0 1 0
Heat_27H_1 27 9 0 0 1 0
Heat_27H_2 27 9 0 0 1 0
Heat_27H_3 27 9 0 0 1 0
Salt_3H_1 3 10 0 0 0 1
Salt_3H_2 3 10 0 0 0 1
Salt_3H_3 3 10 0 0 0 1
Salt_9H_1 9 11 0 0 0 1
Salt_9H_2 9 11 0 0 0 1
Salt_9H_3 9 11 0 0 0 1
Salt_27H_1 27 12 0 0 0 1
Salt_27H_2 27 12 0 0 0 1
Salt_27H_3 27 12 0 0 0 1
class(edesign.abiotic)
[1] "matrix"
data.abiotic[1,]
Control_3H_1 Control_3H_2 Control_3H_3 Control_9H_1 Control_9H_2
STMDF90 0.1373571 -0.3653065 -0.1532945 0.4475454 0.2874768
Control_9H_3 Control_27H_1 Control_27H_2 Control_27H_3 Cold_3H_1
STMDF90 0.2488187 0.1793259 0.1279931 -0.1173468 1.056555
Cold_3H_2 Cold_3H_3 Cold_9H_1 Cold_9H_2 Cold_9H_3 Cold_27H_1 Cold_27H_2
STMDF90 0.3948921 0.3884203 0.8910656 0.9550419 0.8122386 0.9498117 0.7211795
Cold_27H_3 Heat_3H_1 Heat_3H_2 Heat_3H_3 Heat_9H_1 Heat_9H_2
STMDF90 0.6432118 0.1977278 0.08401729 -0.1252850 0.3500279 0.05356246
Heat_9H_3 Heat_27H_1 Heat_27H_2 Heat_27H_3 Salt_3H_1 Salt_3H_2
STMDF90 -0.05703404 0.1425164 0.2824874 0.03085787 0.2681767 0.06403428
Salt_3H_3 Salt_9H_1 Salt_9H_2 Salt_9H_3 Salt_27H_1 Salt_27H_2
STMDF90 0.1757832 0.1050698 0.6643922 0.5167695 0.4754657 0.3387966
Salt_27H_3
STMDF90 0.3596021
> class(data.abiotic)
[1] "data.frame"
#簡単のため、データをちょっといじらせてもらいます。
edesign <- edesign.abiotic[1:18, 1:4]
edesign
Time Replicate Control Cold
Control_3H_1 3 1 1 0
Control_3H_2 3 1 1 0
Control_3H_3 3 1 1 0
Control_9H_1 9 2 1 0
Control_9H_2 9 2 1 0
Control_9H_3 9 2 1 0
Control_27H_1 27 3 1 0
Control_27H_2 27 3 1 0
Control_27H_3 27 3 1 0
Cold_3H_1 3 4 0 1
Cold_3H_2 3 4 0 1
Cold_3H_3 3 4 0 1
Cold_9H_1 9 5 0 1
Cold_9H_2 9 5 0 1
Cold_9H_3 9 5 0 1
Cold_27H_1 27 6 0 1
Cold_27H_2 27 6 0 1
Cold_27H_3 27 6 0 1
#これで、このデータの変数は、「時間」と「Treatment」の二つになった。同様にして
data <- data.abiotic[,1:18]
colname(data)
[1] "Control_3H_1" "Control_3H_2" "Control_3H_3" "Control_9H_1"
[5] "Control_9H_2" "Control_9H_3" "Control_27H_1" "Control_27H_2"
[9] "Control_27H_3" "Cold_3H_1" "Cold_3H_2" "Cold_3H_3"
[13] "Cold_9H_1" "Cold_9H_2" "Cold_9H_3" "Cold_27H_1"
[17] "Cold_27H_2" "Cold_27H_3"
design <- make.design.matrix(edesign, degree = 2)
#"This example has three time points , so we can consider up to a quadratic regression model(degree = 2).Lager number of time points woudld potentially allow a higher polynominal degree.
class(design)
[1] "list"
design
$dis
ColdvsControl Time TimexCold Time2 Time2xCold
Control_3H_1 0 3 0 9 0
Control_3H_2 0 3 0 9 0
Control_3H_3 0 3 0 9 0
Control_9H_1 0 9 0 81 0
Control_9H_2 0 9 0 81 0
Control_9H_3 0 9 0 81 0
Control_27H_1 0 27 0 729 0
Control_27H_2 0 27 0 729 0
Control_27H_3 0 27 0 729 0
Cold_3H_1 1 3 3 9 9
Cold_3H_2 1 3 3 9 9
Cold_3H_3 1 3 3 9 9
Cold_9H_1 1 9 9 81 81
Cold_9H_2 1 9 9 81 81
Cold_9H_3 1 9 9 81 81
Cold_27H_1 1 27 27 729 729
Cold_27H_2 1 27 27 729 729
Cold_27H_3 1 27 27 729 729
$groups.vector
[1] "ColdvsControl" "Control" "ColdvsControl" "Control"
[5] "ColdvsControl"
$edesign
Time Replicate Control Cold
Control_3H_1 3 1 1 0
Control_3H_2 3 1 1 0
Control_3H_3 3 1 1 0
Control_9H_1 9 2 1 0
Control_9H_2 9 2 1 0
Control_9H_3 9 2 1 0
Control_27H_1 27 3 1 0
Control_27H_2 27 3 1 0
Control_27H_3 27 3 1 0
Cold_3H_1 3 4 0 1
Cold_3H_2 3 4 0 1
Cold_3H_3 3 4 0 1
Cold_9H_1 9 5 0 1
Cold_9H_2 9 5 0 1
Cold_9H_3 9 5 0 1
Cold_27H_1 27 6 0 1
Cold_27H_2 27 6 0 1
Cold_27H_3 27 6 0 1
fit <- p.vector (data, design, Q=0.05,MT.adjust = "BH",min.obs = 3)
#min.obs : genes with less than this number of true numerical values will be excluded from the analysi.Default is 3(minimun value for a quadratic fit)
tstep <- T.fit(fit, step.method = "backward", alfa = 0.05)
#次に発現変動遺伝子を得ます。
sigs <- get.siggenes(tstep, rsq = 0.6, vars ="groups")
class(sigs)
[1] "list"
names(sigs)
[1] "sig.genes" "summary"
Control ColdvsControl
1 STMDF90 STMDF90
2 STMIA38 STMJH42
3 STMEQ29 STMDE66
4 STMEL85 STMHZ45
5 STMGU57 STMGL58
6 STMHK85 STMIF71
7 STMHJ39 STMIA38
8 STMGB57 STMEQ29
9 STMIT31 STMDW06
10 STMEY09 STMEL85
11 STMHY91 STMEG74
12 STMHS90 STMGU57
13 STMCU02 STMDV94
14 STMGB35 STMHK85
15 STMIH90 STMDV87
16 STMCF08 STMID12
17 STMDI90 STMCV66
18 STMIG08 STMGH56
19 STMCX95 STMEJ16
20 STMCO80 STMCD46
21 STMEM80 STMJE19
22 STMCF73 STMHJ39
23 STMGC06 STMJH69
24 STMEZ88 STMGB57
25 STMER65 STMIT31
26 STMHL59 STMEZ42
27 STMIK50 STMHN16
28 STMEY29 STMEY09
29 STMDT77 STMCE01
30 STMDM56 STMDG64
31 STMDM29 STMIY82
32 STMCH02 STMFB85
33 STMCS44 STMHH10
34 STMJJ85 STMGQ20
35 STMGG37 STMCY10
36 STMCH67 STMHV34
37 STMJF53 STMHY91
38 STMIQ37 STMIV44
39 STMJN55 STMDJ72
40 STMCO78 STMHS90
41 STMCM86 STMJN05
42 STMDF13 STMCH90
43 STMET96 STMIX07
44 STMIT39 STMEF65
45 STMCK87 STMCA27
46 STMIB81 STMEG36
47 STMCU87 STMGZ67
48 STMHN19 STMEB60
49 STMJI65 STMIH67
50 STMED61 STMII96
51 STMCI43 STMCU02
52 STMHU96 STMEU58
53 STMCH79 STMGM14
54 STMDU84 STMDJ03
55 STMGO62 STMGB35
56 STMIO93 STMEH38
57 STMCB61 STMGP81
58 STMEB31 STMCS39
59 STMIW42 STMCD51
60 STMGT19 STMHY77
61 STMEG09 STMGH21
62 STMCB20 STMJP11
63 STMIW62 STMIH90
64 STMHF88 STMCE67
65 STMIX47 STMIA37
66 STMCF08
67 STMHR19
68 STMHK44
69 STMDI90
70 STMIG08
71 STMEA14
72 STMCX95
73 STMCR17
74 STMCO80
75 STMDF62
76 STMDU07
77 STMDF61
78 STMIR22
79 STMIS03
80 STMEM80
81 STMCF73
82 STMJJ41
83 STMIA39
84 STMGC06
85 STMIO60
86 STMEZ88
87 STMJJ12
-----省略します------
#まずは時間に関して有意変化がありかつ、controlvscolでも有意なSTMDF90について
STMDF90 <- data[rownames(data) == "STMDF90", ]
png("STMDF90.png")
PlotGroups(STMDF90, edesign = edesign, show.fit = T, dis = design$dis, groups.vector = design$groups.vector,main = "SDMF90")
dev.off()
#see.genes(9 performs a cluster analysis to group genes by similar profiles.The resulting clusters are then plotted in two fashions:as experiment-wide expression profiles and as by-groups profies.
pdf("ColdsvsControl.pdf")
see.genes(sigs$sig.genes$ColdvsControl, main = "ColdvsControl", dis =design$dis, cluster.method="kmeans" ,cluster.data = 1, k = 9)
dev.off()
png("ColdsvsControl.png")
par(mfrow = c(9,9))
see.genes(sigs$sig.genes$ColdvsControl, main = "ColdvsControl", show.fit = T, dis =design$dis, cluster.method="kmeans" ,cluster.data = 1, k = 9)
dev.off()
png("ColdsvsControl.test.png")
s
dev.off()
biocLite("maSigPro")
library(maSigPro)
data(edesign.abiotic)
data(data.abiotic)
edesign.abiotic
Time Replicate Control Cold Heat Salt
Control_3H_1 3 1 1 0 0 0
Control_3H_2 3 1 1 0 0 0
Control_3H_3 3 1 1 0 0 0
Control_9H_1 9 2 1 0 0 0
Control_9H_2 9 2 1 0 0 0
Control_9H_3 9 2 1 0 0 0
Control_27H_1 27 3 1 0 0 0
Control_27H_2 27 3 1 0 0 0
Control_27H_3 27 3 1 0 0 0
Cold_3H_1 3 4 0 1 0 0
Cold_3H_2 3 4 0 1 0 0
Cold_3H_3 3 4 0 1 0 0
Cold_9H_1 9 5 0 1 0 0
Cold_9H_2 9 5 0 1 0 0
Cold_9H_3 9 5 0 1 0 0
Cold_27H_1 27 6 0 1 0 0
Cold_27H_2 27 6 0 1 0 0
Cold_27H_3 27 6 0 1 0 0
Heat_3H_1 3 7 0 0 1 0
Heat_3H_2 3 7 0 0 1 0
Heat_3H_3 3 7 0 0 1 0
Heat_9H_1 9 8 0 0 1 0
Heat_9H_2 9 8 0 0 1 0
Heat_9H_3 9 8 0 0 1 0
Heat_27H_1 27 9 0 0 1 0
Heat_27H_2 27 9 0 0 1 0
Heat_27H_3 27 9 0 0 1 0
Salt_3H_1 3 10 0 0 0 1
Salt_3H_2 3 10 0 0 0 1
Salt_3H_3 3 10 0 0 0 1
Salt_9H_1 9 11 0 0 0 1
Salt_9H_2 9 11 0 0 0 1
Salt_9H_3 9 11 0 0 0 1
Salt_27H_1 27 12 0 0 0 1
Salt_27H_2 27 12 0 0 0 1
Salt_27H_3 27 12 0 0 0 1
class(edesign.abiotic)
[1] "matrix"
data.abiotic[1,]
Control_3H_1 Control_3H_2 Control_3H_3 Control_9H_1 Control_9H_2
STMDF90 0.1373571 -0.3653065 -0.1532945 0.4475454 0.2874768
Control_9H_3 Control_27H_1 Control_27H_2 Control_27H_3 Cold_3H_1
STMDF90 0.2488187 0.1793259 0.1279931 -0.1173468 1.056555
Cold_3H_2 Cold_3H_3 Cold_9H_1 Cold_9H_2 Cold_9H_3 Cold_27H_1 Cold_27H_2
STMDF90 0.3948921 0.3884203 0.8910656 0.9550419 0.8122386 0.9498117 0.7211795
Cold_27H_3 Heat_3H_1 Heat_3H_2 Heat_3H_3 Heat_9H_1 Heat_9H_2
STMDF90 0.6432118 0.1977278 0.08401729 -0.1252850 0.3500279 0.05356246
Heat_9H_3 Heat_27H_1 Heat_27H_2 Heat_27H_3 Salt_3H_1 Salt_3H_2
STMDF90 -0.05703404 0.1425164 0.2824874 0.03085787 0.2681767 0.06403428
Salt_3H_3 Salt_9H_1 Salt_9H_2 Salt_9H_3 Salt_27H_1 Salt_27H_2
STMDF90 0.1757832 0.1050698 0.6643922 0.5167695 0.4754657 0.3387966
Salt_27H_3
STMDF90 0.3596021
> class(data.abiotic)
[1] "data.frame"
#簡単のため、データをちょっといじらせてもらいます。
edesign <- edesign.abiotic[1:18, 1:4]
edesign
Time Replicate Control Cold
Control_3H_1 3 1 1 0
Control_3H_2 3 1 1 0
Control_3H_3 3 1 1 0
Control_9H_1 9 2 1 0
Control_9H_2 9 2 1 0
Control_9H_3 9 2 1 0
Control_27H_1 27 3 1 0
Control_27H_2 27 3 1 0
Control_27H_3 27 3 1 0
Cold_3H_1 3 4 0 1
Cold_3H_2 3 4 0 1
Cold_3H_3 3 4 0 1
Cold_9H_1 9 5 0 1
Cold_9H_2 9 5 0 1
Cold_9H_3 9 5 0 1
Cold_27H_1 27 6 0 1
Cold_27H_2 27 6 0 1
Cold_27H_3 27 6 0 1
#これで、このデータの変数は、「時間」と「Treatment」の二つになった。同様にして
data <- data.abiotic[,1:18]
colname(data)
[1] "Control_3H_1" "Control_3H_2" "Control_3H_3" "Control_9H_1"
[5] "Control_9H_2" "Control_9H_3" "Control_27H_1" "Control_27H_2"
[9] "Control_27H_3" "Cold_3H_1" "Cold_3H_2" "Cold_3H_3"
[13] "Cold_9H_1" "Cold_9H_2" "Cold_9H_3" "Cold_27H_1"
[17] "Cold_27H_2" "Cold_27H_3"
design <- make.design.matrix(edesign, degree = 2)
#"This example has three time points , so we can consider up to a quadratic regression model(degree = 2).Lager number of time points woudld potentially allow a higher polynominal degree.
class(design)
[1] "list"
design
$dis
ColdvsControl Time TimexCold Time2 Time2xCold
Control_3H_1 0 3 0 9 0
Control_3H_2 0 3 0 9 0
Control_3H_3 0 3 0 9 0
Control_9H_1 0 9 0 81 0
Control_9H_2 0 9 0 81 0
Control_9H_3 0 9 0 81 0
Control_27H_1 0 27 0 729 0
Control_27H_2 0 27 0 729 0
Control_27H_3 0 27 0 729 0
Cold_3H_1 1 3 3 9 9
Cold_3H_2 1 3 3 9 9
Cold_3H_3 1 3 3 9 9
Cold_9H_1 1 9 9 81 81
Cold_9H_2 1 9 9 81 81
Cold_9H_3 1 9 9 81 81
Cold_27H_1 1 27 27 729 729
Cold_27H_2 1 27 27 729 729
Cold_27H_3 1 27 27 729 729
$groups.vector
[1] "ColdvsControl" "Control" "ColdvsControl" "Control"
[5] "ColdvsControl"
$edesign
Time Replicate Control Cold
Control_3H_1 3 1 1 0
Control_3H_2 3 1 1 0
Control_3H_3 3 1 1 0
Control_9H_1 9 2 1 0
Control_9H_2 9 2 1 0
Control_9H_3 9 2 1 0
Control_27H_1 27 3 1 0
Control_27H_2 27 3 1 0
Control_27H_3 27 3 1 0
Cold_3H_1 3 4 0 1
Cold_3H_2 3 4 0 1
Cold_3H_3 3 4 0 1
Cold_9H_1 9 5 0 1
Cold_9H_2 9 5 0 1
Cold_9H_3 9 5 0 1
Cold_27H_1 27 6 0 1
Cold_27H_2 27 6 0 1
Cold_27H_3 27 6 0 1
fit <- p.vector (data, design, Q=0.05,MT.adjust = "BH",min.obs = 3)
#min.obs : genes with less than this number of true numerical values will be excluded from the analysi.Default is 3(minimun value for a quadratic fit)
tstep <- T.fit(fit, step.method = "backward", alfa = 0.05)
#次に発現変動遺伝子を得ます。
sigs <- get.siggenes(tstep, rsq = 0.6, vars ="groups")
class(sigs)
[1] "list"
names(sigs)
[1] "sig.genes" "summary"
Control ColdvsControl
1 STMDF90 STMDF90
2 STMIA38 STMJH42
3 STMEQ29 STMDE66
4 STMEL85 STMHZ45
5 STMGU57 STMGL58
6 STMHK85 STMIF71
7 STMHJ39 STMIA38
8 STMGB57 STMEQ29
9 STMIT31 STMDW06
10 STMEY09 STMEL85
11 STMHY91 STMEG74
12 STMHS90 STMGU57
13 STMCU02 STMDV94
14 STMGB35 STMHK85
15 STMIH90 STMDV87
16 STMCF08 STMID12
17 STMDI90 STMCV66
18 STMIG08 STMGH56
19 STMCX95 STMEJ16
20 STMCO80 STMCD46
21 STMEM80 STMJE19
22 STMCF73 STMHJ39
23 STMGC06 STMJH69
24 STMEZ88 STMGB57
25 STMER65 STMIT31
26 STMHL59 STMEZ42
27 STMIK50 STMHN16
28 STMEY29 STMEY09
29 STMDT77 STMCE01
30 STMDM56 STMDG64
31 STMDM29 STMIY82
32 STMCH02 STMFB85
33 STMCS44 STMHH10
34 STMJJ85 STMGQ20
35 STMGG37 STMCY10
36 STMCH67 STMHV34
37 STMJF53 STMHY91
38 STMIQ37 STMIV44
39 STMJN55 STMDJ72
40 STMCO78 STMHS90
41 STMCM86 STMJN05
42 STMDF13 STMCH90
43 STMET96 STMIX07
44 STMIT39 STMEF65
45 STMCK87 STMCA27
46 STMIB81 STMEG36
47 STMCU87 STMGZ67
48 STMHN19 STMEB60
49 STMJI65 STMIH67
50 STMED61 STMII96
51 STMCI43 STMCU02
52 STMHU96 STMEU58
53 STMCH79 STMGM14
54 STMDU84 STMDJ03
55 STMGO62 STMGB35
56 STMIO93 STMEH38
57 STMCB61 STMGP81
58 STMEB31 STMCS39
59 STMIW42 STMCD51
60 STMGT19 STMHY77
61 STMEG09 STMGH21
62 STMCB20 STMJP11
63 STMIW62 STMIH90
64 STMHF88 STMCE67
65 STMIX47 STMIA37
66 STMCF08
67 STMHR19
68 STMHK44
69 STMDI90
70 STMIG08
71 STMEA14
72 STMCX95
73 STMCR17
74 STMCO80
75 STMDF62
76 STMDU07
77 STMDF61
78 STMIR22
79 STMIS03
80 STMEM80
81 STMCF73
82 STMJJ41
83 STMIA39
84 STMGC06
85 STMIO60
86 STMEZ88
87 STMJJ12
-----省略します------
#まずは時間に関して有意変化がありかつ、controlvscolでも有意なSTMDF90について
STMDF90 <- data[rownames(data) == "STMDF90", ]
png("STMDF90.png")
PlotGroups(STMDF90, edesign = edesign, show.fit = T, dis = design$dis, groups.vector = design$groups.vector,main = "SDMF90")
dev.off()
#see.genes(9 performs a cluster analysis to group genes by similar profiles.The resulting clusters are then plotted in two fashions:as experiment-wide expression profiles and as by-groups profies.
pdf("ColdsvsControl.pdf")
see.genes(sigs$sig.genes$ColdvsControl, main = "ColdvsControl", dis =design$dis, cluster.method="kmeans" ,cluster.data = 1, k = 9)
dev.off()
png("ColdsvsControl.png")
par(mfrow = c(9,9))
see.genes(sigs$sig.genes$ColdvsControl, main = "ColdvsControl", show.fit = T, dis =design$dis, cluster.method="kmeans" ,cluster.data = 1, k = 9)
dev.off()
png("ColdsvsControl.test.png")
s
dev.off()
マイクロアレイデータの描写のモデルを作ってみたよん♪
$ R
arrayModel <- matrix(rep(0:1,121), 11, 11)
arrayModel
[,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10] [,11]
[1,] 0 1 0 1 0 1 0 1 0 1 0
[2,] 1 0 1 0 1 0 1 0 1 0 1
[3,] 0 1 0 1 0 1 0 1 0 1 0
[4,] 1 0 1 0 1 0 1 0 1 0 1
[5,] 0 1 0 1 0 1 0 1 0 1 0
[6,] 1 0 1 0 1 0 1 0 1 0 1
[7,] 0 1 0 1 0 1 0 1 0 1 0
[8,] 1 0 1 0 1 0 1 0 1 0 1
[9,] 0 1 0 1 0 1 0 1 0 1 0
[10,] 1 0 1 0 1 0 1 0 1 0 1
[11,] 0 1 0 1 0 1 0 1 0 1 0
png("110211_R.matrix.png")
par(mfrow = c(1,3))
image(arrayModel, col=c(1,0),main="arrayModel matrix")
image(arrayModel, col=c(0,1),main="arrayModel matrix")
image(arrayModel, col=c(2,3),main="arrayModel matrix")
dev.off()
よりマイクロアレイのシチュエーションに近付けてみるよん!!
png("110211_R.matrix.2.png")
arrayModel <- matrix(rep(0:7,121), 11, 11)
image(arrayModel, col=c(2,3,8,7,5,11,9),main="arrayModel matrix")
dev.off()
png("110211_R.matrix.3.png")
arrayModel <- matrix(rep(0:111,111*111), 7, 111)
image(arrayModel, col=c(2,3,8,7,5,11,9),main="arrayModel matrix")
dev.off()
マイクロアレイのデータは本質的には、このようなマトリックスデータである。但し、各々のスポットは連続変数であるため、今回のモデルよりも美しい絵になるわけである。
arrayModel <- matrix(rep(0:1,121), 11, 11)
arrayModel
[,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10] [,11]
[1,] 0 1 0 1 0 1 0 1 0 1 0
[2,] 1 0 1 0 1 0 1 0 1 0 1
[3,] 0 1 0 1 0 1 0 1 0 1 0
[4,] 1 0 1 0 1 0 1 0 1 0 1
[5,] 0 1 0 1 0 1 0 1 0 1 0
[6,] 1 0 1 0 1 0 1 0 1 0 1
[7,] 0 1 0 1 0 1 0 1 0 1 0
[8,] 1 0 1 0 1 0 1 0 1 0 1
[9,] 0 1 0 1 0 1 0 1 0 1 0
[10,] 1 0 1 0 1 0 1 0 1 0 1
[11,] 0 1 0 1 0 1 0 1 0 1 0
png("110211_R.matrix.png")
par(mfrow = c(1,3))
image(arrayModel, col=c(1,0),main="arrayModel matrix")
image(arrayModel, col=c(0,1),main="arrayModel matrix")
image(arrayModel, col=c(2,3),main="arrayModel matrix")
dev.off()
よりマイクロアレイのシチュエーションに近付けてみるよん!!
png("110211_R.matrix.2.png")
arrayModel <- matrix(rep(0:7,121), 11, 11)
image(arrayModel, col=c(2,3,8,7,5,11,9),main="arrayModel matrix")
dev.off()
png("110211_R.matrix.3.png")
arrayModel <- matrix(rep(0:111,111*111), 7, 111)
image(arrayModel, col=c(2,3,8,7,5,11,9),main="arrayModel matrix")
dev.off()
マイクロアレイのデータは本質的には、このようなマトリックスデータである。但し、各々のスポットは連続変数であるため、今回のモデルよりも美しい絵になるわけである。
登録:
コメント (Atom)