データはNBPSeqというパッケージに付属のarabを用いました。
これはシロイヌナズナ(Arabidopsis)のmutantとコントロールの二群から各々3つずつサンプリングされ、Illumina Genome Analyzer(http://www.illumina.com)によりシーケンスされたものです。
###データの読み込みと簡単な評価##
#NBPSeqをインストール&ロード
source("http://bioconductor.org/biocLite.R")
biocLite("NBPSeq")
library(NBPSeq)
#arabオブジェクトを生成
data(arab)
#arabの中身を頭出し
> head(arab)
mock1 mock2 mock3 hrcc1 hrcc2 hrcc3
AT1G01010 35 77 40 46 64 60
AT1G01020 43 45 32 43 39 49
AT1G01030 16 24 26 27 35 20
AT1G01040 72 43 64 66 25 90
AT1G01050 49 78 90 67 45 60
AT1G01060 0 15 2 0 21 8
#NGSのデータ特有の整数値が格納されていることが確認できる。
#arabオブジェクトの構造(各カラムのモード等)を表示
> str(arab)
int [1:26222, 1:6] 35 43 16 72 49 0 16 170 291 113 ...
- attr(*, "dimnames")=List of 2
..$ : chr [1:26222] "AT1G01010" "AT1G01020" "AT1G01030" "AT1G01040" ...
..$ : chr [1:6] "mock1" "mock2" "mock3" "hrcc1" ...
####データの可視化により定性的な評価を行う###
#サンプル間のすべての組み合わせに対してscatter plotを行い、technical duplicateとbiological duplicateのばらつきを評価する。
pairs(arab, log="xy", pch=16, cex=.3)
#hとmの各々について行の平均値を計算する
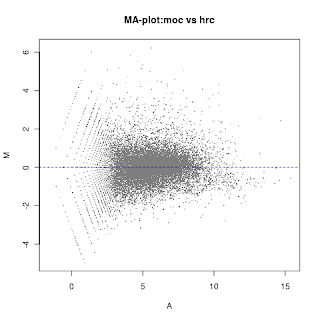
mean_m <- apply(arab, 1, function(x){mean(x[1:3])}) mean_h <- apply(arab, 1, function(x){mean(x[4:6])}) #MAプロットを行うために、M (log fold-change)とA (log intensity)を求める。 M <- log2(mean_h) - log2(mean_m) A <- 1/2 * (log2(mean_h) + log2(mean_m)) #MAプロットを行う plot(A, M, pch=16, cex=0.3, col="gray50",main="MA-plot:moc vs hrc) #M=0の高さに水平線を引く。 abline(h=0, col=4, lty=2)
###5倍以上の発現変動があった遺伝子をピックアップする。###
#これまでの計算結果をひとつのテーブルにまとめる
arab_summary <- data.frame(arab, mean_m, mean_h, M, A) #各行で発現変動があったかどうかを調べて二値で表す。(RPKMなどの手法で正規化を行っていない生データに対して解析を行うのは乱暴であるが。。) > head(M >= log2(5))
AT1G01010 AT1G01020 AT1G01030 AT1G01040 AT1G01050 AT1G01060
FALSE FALSE FALSE FALSE FALSE FALSE
#5倍以上の発現変動があった遺伝子を抽出し、新たなテーブルを作成
five_fold <- subset(arab_summary, arab_summary$M >= log2(5))
#MA-plotを描写して、5倍異常の発現変動があった遺伝子を赤くマーキングする
plot(arab_summary$A, arab_summary$M, pch=16, cex=0.3, col="gray50")
points(five_fold$A, five_fold$M, col="red", pch=16, cex=0.4)
#倍数変化の手法だと、発現値の低い遺伝子(ばらつきの大きな傾向にあります)にたくさんの偽陽性が含まれて、
#発現値の高い遺伝子(ばらつきが小さい傾向にあります)に偽陰性がたくさんふくまれてしまいます。
#しっかりデータ全体にたいして正規化を行った後、t-testなどでp値を用いた検定を行い、その両者の共通項を探しにいくのが無難かと思われます。

